Surveillance de l'ARN messager — Wikipédia
Les mécanismes de surveillance de l'ARNm sont des voies utilisées par les organismes pour assurer la fidélité et la qualité des molécules d'ARN messagers. Il existe un grand nombre de mécanismes différents. Ils interviennent à des étapes variées de la vie d'un ARNm pour détecter et dégrader les brins d'ARNm qui présentent des défauts de synthèse, des mutations ou des dégradations.
La traduction des ARNm défectueux peut en effet conduire à des effets très délétères pour l'organisme. Les protéines anormales qui résultent de la traduction de ces ARN ont en effet tendance à former des agrégats insolubles. L'accumulation de ces agrégats conduit à la mort cellulaire et à la formation de dépôts amyloïdes dans les tissus.
Vue d'ensemble
[modifier | modifier le code]La traduction de l'ARN messager en protéines est une étape vitale de la théorie fondamentale de la biologie moléculaire. Les molécules d'ARNm peuvent cependant comporter des erreurs dues à des mutations dans le génomes, au manque de fidélité de l'ARN polymérase lors de la transcription, elles peuvent également subir des coupures nucléolytiques spontanées ou induites par des ribonucléases. Il peut en résulter des ARNm incorrects ou incomplets dont la traduction pourrait se répercuter sur la qualité des protéines[1] correspondantes.
Les mécanismes de surveillance de l'ARN permettent d'assurer le contrôle qualité des ARNm[2]. Cela est rendu possible en général par le marquage des ARNm aberrants, qui les oriente vers les voies de dégradation par des nucléases endogènes[3].
La surveillance de l'ARNm a été mise en évidence chez les bactéries et les eukaryotes. Chez ces derniers, ces mécanismes prennent place dans le noyau cellulaire et dans le cytoplasme[4]. Les contrôles intervenant dans le noyau assurent la fidélité de la molécule d'ARNm, ceux qui interviennent dans le cytoplasmes vérifient l'absence de codons stop prématurés[3],[4].
Chez les eucaryotes, le mécanisme de recrutement du ribosome lors du démarrage de la traduction constitue un premier niveau de surveillance de l'intégrité de l'ARNm. En effet pour que le ribosome puisse être recruté, il est nécessaire que l'ARNm soit sous forme de pseudo-cercle, avec la queue poly(A) en 3' fixée à la PABP, la coiffe en 5' fixée au facteur de traduction eIF4E, les deux protéines interagissant avec le facteur eIF4G[5]. Ce mécanisme assure avant de démarrer la traduction que l'ARNm est intact et complet, avec une coiffe 5' et une queue poly(A) en 3'. Ainsi, par exemple, les ARNm clivés ou incomplets ne sont pas traduits.

Un mécanisme différent mais de finalité similaire existe chez les bactéries : la trans-traduction par l'ARNtm.
Trois mécanismes de surveillance plus spécifiques ont ensuite été identifiés : Le Nonsense Mediated mRNA Decay (NMD), le Nonstop Mediated mRNA decay (NSD) et le No-Go Mediated mRNA Decay (NGD).
Nonsense Mediated mRNA Decay
[modifier | modifier le code]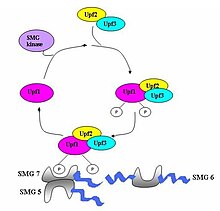
Le Nonsense Mediated decay permet la dégradation des ARN messagers qui contiennent des codons stop prématurés. Ces codons peuvent être présents dans l'ADN à cause d'une mutation, ou apparaître dans l'ARN lors de la transcription ou de l'une des étapes consécutives[6],[7]. Sans le mécanisme de NMD, ces ARNm donneraient naissance à des protéines tronquées potentiellement dangereuses pour l'organisme[8]. 30 % des maladies héréditaires sont en réalité dues à des codons STOP prématurés, ce qui atteste l'importance vitale du NMD pour la survie des organismes[9],[10].
Mécanisme chez les mammifères
[modifier | modifier le code]
Le Nonsense-Mediated Decay repose sur le fait qu'un « vrai » codon stop est toujours situé dans le dernier exon de l'ARN pré-messager[11]. Il a ainsi été montré que ce mécanisme ne fonctionne pas pour les gènes qui ne contiennent pas d'introns (Histone H4, Hsp70, récepteur à la mélanocortine 4…)[8]. Lors de l'épissage, des complexes appelés complexes de jonction d'exons (Exon junction complex ou EJCs) sont déposés à 20-24 nucléotides en amont des jonctions exon-exon[12],[13].
Si le codon stop est bien situé sur le dernier exon, il ne reste normalement pas d'EJC sur l'ARNm lorsque le ribosome l'atteint. Dans le cas contraire, les facteurs de traduction eRF1 et eRF3 interagissent avec les EJC encore présents sur l'ARNm ce qui trahit la présence d'un codon stop prématuré[14]. Cette interaction conduit au marquage de l'ARNm par des facteurs favorisant sa dégradation par les nucléases de la cellule[11],[14].
Contournement du NMD
[modifier | modifier le code]Les brins d'ARNm avec des mutations non-sens sont généralement destinées à être dégradée par la voie NMD. Cependant, il a été observé que certaines molécules avec un codon stop prématuré peuvent éviter la détection et la dégradation[15]. En général, le codon stop apparaît très tôt sur l'ARNm, à proximité de l'AUG initial. Cela est en apparence en contradiction avec le modèle actuellement accepté pour le NMD[16].
Ce phénomène a été montré avec les β-globulines. Les ARNm codant les β-globulines qui contiennent une mutation dans le premier exon du gène sont particulièrement stables vis-à-vis du NMD. Le mécanisme exact d'évitement de la détection n'est pas encore connu, mais il a été suggéré que les poly(A)-binding protein (PABP) jouent un rôle dans cette stabilité[17]. D'autres études ont montré que la présence de cette protéine près des codons stop prématurés augmente généralement la stabilité des ARNm face au NMD, y compris pour des ARNm autres que celui de la β-globuline[15]. Le modèle actuel de compréhension du NMD pourrait donc avoir besoin d'être actualisé dans le cadre d'études futures.
Nonstop Mediated mRNA Decay
[modifier | modifier le code]Vue d'ensemble
[modifier | modifier le code]
Le Nonstop Mediated decay (NSD) entre en jeu dans la détection et la dégradation des ARNm qui ne possèdent pas de codon stop[19],[20]. Ces ARNm peuvent résulter d'une adénylation en 3' prématurée ou d'un signal de polyadénylation cryptique présent dans la région codante d'un gène[21]. Cette absence de codon stop est délétère pour la cellule : quand le ribosome parvient à la queue 3' Poly-A de l'ARNm, il s'immobilise et ne peut pas éjecter l'ARNm[22]. Il n'est donc plus possible pour ces ribosomes de participer à la synthèse des protéines. Le nonstop mediated decay intervient afin de détacher les ribosomes immobilisés de marquer les ARNm défectueux pour qu'ils soient dégradés par des nucléases. Deux voies de nonstop mediated decay ont été identifiées et agissent probablement conjointement[19],[20].
Voie Ski7
[modifier | modifier le code]Cette voie est active quand la protéine Ski7 est présente dans la cellule. Cette protéine se lie au site A du ribosome quand il est vide. Cela a pour conséquence l'éjection du ribosome, ce qui le rend disponible pour la traduction d'autres protéines. Ski7 reste attachée à l'ARNm sans codon stop, et sert d'élément de reconnaissance pour l'exosome, entrainant la déadénylation de l'ARNm puis sa dégradation de 3' vers 5'[19],[20].
Voie indépendante de Ski7
[modifier | modifier le code]L'autre voie de NSD a été observée chez la levure. En l'absence de Ski7, l'arrivée du ribosome dans la queue poly-A entraîne la perte des protéines PABP qui y sont fixées. Sans ces protéines, le brin d'ARNm perd sa coiffe de protection 5' m7G. Des exonucléases 5'-3' endogènes comme Xrn1 peuvent alors dégrader rapidement l'ARNm dépourvu de codon stop[20].
No-Go Decay
[modifier | modifier le code]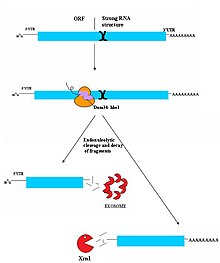
Le No-Go decay (NGD) est le mécanisme de surveillance découvert le plus récemment[23]. Il n'est donc pas encore totalement compris. Il semblerait que les cibles du NGD soient des brins d'ARNm sur lesquels les ribosomes se sont arrêtés pendant la traduction. De nombreuses causes peuvent entrainer un arrêt du ribosome, comme une forte structure secondaire de l'ARN[23]. Les protéines Dom34/Hbs1 se fixent probablement près du site A des ribosomes immobilisés et facilitent le recyclage des complexes[24].Dans certains cas, l'ARNm est aussi coupé près de site d'immobilisation, bien que la nature de l'endonucléase qui entre en jeu est sujette à débat. Les molécules d'ARN fragmentées seront alors dégradées par l'exosome de 3' vers 5', ou par Xrn1 de 5' vers 3'[23]. Le mécanisme conduisant au détachement du ribosome n'est pas connu, mais la protéine Hbs1 possède une forte similarité de structure avec Ski7 et pourrait jouer dans la NGD un rôle équivalent à celui de Ski7 dans la NSD[6],[25].
Évolution des mécanismes de surveillance de l'ARNm
[modifier | modifier le code]
Il est possible de déterminer l'histoire évolutionnaire de ces mécanismes en observant la conservation des protéines impliquées dans chaque mécanisme. Des recherches BLAST étendues ont été réalisées pour déterminer la prévalence de ces protéines chez différents types d'organismes. Par exemple, la protéine de le NGD Hbs1 et la protéine du NSD eRF3 ne sont retrouvés que chez les eucaryotes. En revanche, la protéine du NGD Dom34 est commune aux eucaryotes et aux archées. Ceci suggère que le NGD a été le premier mécanisme de surveillance à apparaître dans l'évolution. La protéine Ski7 du NSD est restreinte aux levures, laissant penser que son apparition est récente[26].
Références
[modifier | modifier le code]- (en) Cet article est partiellement ou en totalité issu de l’article de Wikipédia en anglais intitulé « mRNA surveillance » (voir la liste des auteurs).
- Amrani, N., Sachs, M.S. and Jacobson, A. (2006) Early nonsense:mRNA decay solves a translational problem. Nat. Mol. Cell.Biol. 7, 415–425.
- Moore, M.J. (2005) From birth to death: the complex lives of eukaryotic mRNAs. Science 309, 1514–1518.
- Amrani, R. Ganesan, S. Kervestin, D.A. Mangus, S. Ghosh and A. Jacobson, A faux 3'-UTR promotes aberrant termination and triggers nonsense-mediated mRNA decay, Nature 432 (2004), p. 112–118.
- Fasken, M.B. and Corbett, A.H. (2005) Process or perish: quality control in mRNA biogenesis. Nat. Struct. Mol. Biol. 12, 482–488.
- S. Z. Tarun et A. B. Sachs, « Association of the yeast poly(A) tail binding protein with translation initiation factor eIF-4G », The EMBO journal, vol. 15, , p. 7168–7177 (ISSN 0261-4189, PMID 9003792, PMCID 452544, lire en ligne, consulté le )
- Chang, Y.F., Imam, J.S. and Wilkinson, M.F. (2007) The nonsense-mediated decay RNA surveillance pathway. Biochem. 76, 51–74.
- Rehwinkel, J., Raes, J. and Izaurralde, E. (2006) Nonsense-mediated mRNA decay: target genes and functional diversification of effectors. Trend Biochem. Sci. 31, 639–646.
- Maquat, L.E. (2004) Nonsense-mediated mRNA decay: splicing, translation and mRNP dynamics. Nat. Mol. Cell. Biol. 5, 89–99.
- Holbrook, J.A., Neu-Yilik, G., Hentze, A.E. and Kulozik, M.W. (2004) nonsensemediated decay approaches the clinic. Nat. Genet. 36, 801–808.
- Mendell, J.T., Sharifi, N.A., Meyers, J.L., Martinez-Murillo, F. and Dietz, H.C. (2004) Nonsense surveillance regulates expression of diverse classes of mammalian transcripts and mutes genomic noise. Nat. Genet. 36, 1073–1078.
- Zhang, J., Sun, X., Qian, Y., LaDuca, J.P. and Maquat, L.E. (1998) At least one intron is required for the nonsense-mediated decay of triosephosphate isomerase mRNA: a possible link between nuclear splicing and cytoplasmic translation. Mol. Cell. Biol. 18, 5272–5283.
- Neu-Yilik, G., Gehring, N.H., Thermann, R., Frede, U., Hentze, M.W. and Kulozik, A.E. (2001) Splicing and 30 end formation in the definition of nonsensemediated decay-competent human beta-globin mRNPs. EMBO J. 20, 532–540.
- Behm-Ansmant, I., Gatfield, D., Rehwinkel, J., Hilgers.V., and Izaurralde, J., A conserved role for cytoplasmic poly(A)-binding protein 1 (PABPC1) in nonsense-mediated mRNA decay, EMBO J. 26 (2007), p. 1591–1601.
- Kashima, I., Yamashita, A., Izumi, N., Kataoka, N., Morishita, R., Hoshino, S., Ohno, M., Dreyfuss, G., and Ohno, S. 2006. Binding of a novel SMG-1-Upf1-eRF1-eRF3 complex (SURF) to the exon junction complex triggers Upf1 phosphorylation and nonsensemediated mRNA decay. Genes & Dev. 20: 355–367.
- Inácio, A., Silva, A.L., Pinto, J., Ji, X., Morgado, A., Almeida, F.,Faustino, P., Lavinha, J., Liebhaber, S.A., and Romão, L. 2004. Nonsense mutations in close proximity to the initiation codon fail to trigger full nonsense-mediated mRNA decay. J. Biol. Chem. 279: 32170–32180.
- Silva, A.L., Pereira, F.J., Morgado, A., Kong, J., Martins, R., Faustino, P., Liebhaber, S.A., and Romão, L. 2006. The canonical UPF1-dependent nonsense-mediated mRNA decay is inhibited in transcripts carrying a short open reading frame independent of sequence context. RNA 12: 2160–2170.
- Silva, A.L., Ribeiro, P., Liebhaber, S.A., and Romao, L. (2008). Proximity of the poly(A)-binding protein to a premature termination codon inhibits mammalian nonsense-mediated mRNA decay. RNA. 14: 563-576.
- Garneau NL, Wilusz J, Wilusz CJ: The highways and byways of mRNA decay. Nat Rev Mol Cell Biol 2007, 8:113-126.
- van Hoof, A., Frischmeyer, P. A., Dietz, H. C. & Parker, R. Exosome-mediated recognition and degradation of mRNAs lacking a termination codon. Science 295, 2262–2264 (2002).
- Frischmeyer, P. A. et al. An mRNA surveillance mechanism that eliminates transcripts lacking termination codons. Science 295, 2258–2261 (2002).
- Temperley RJ, Seneca SH, Tonska K, Bartnik E, Bindoff LA, Lightowlers RN, Chrzanowska-Lightowlers ZM (2003) Investigation of a pathogenic mtDNA microdeletion reveals a translation-dependent deadenylation decay pathway in human mitochondria. Hum Mol Genet 12: 2341–2348
- Karzai AW, Roche ED, Sauer RT (2000) The SsrA-SmpB system for protein tagging, directed degradation and ribosome rescue. Nat Struct Biol 7: 449–455
- Doma, M. K. & Parker, R. Endonucleolytic cleavage of eukaryotic mRNAs with stalls in translation elongation. Nature 440, 561–564.(2006).
- Kobayashi K, Kikuno I, Kuroha K, Saito K, Ito K, Ishitani R, Inada T, & Nureki O. Structural basis for mRNA surveillance by archaeal Pelota and GTP-bound EF1α complex. PNAS. 107(41):17575-9. (2010).
- Graille M, Chaillet M, van Tilbeurgh H. Structure of yeast Dom34: a protein related to translation termination factor eRF1 and involved in No-Go decay. J Biol Chem. 2008;283:7145–7154.
- Atkinson GC, Baldauf SL, Hauryliuk V. Evolution of nonstop, no-go and nonsense-mediated mRNA decay and their termination factor-derived components. BMC Evol Biol. 2008 Oct 23;8:290


 French
French Deutsch
Deutsch