Variantes de la PCR — Wikipédia
La polyvalence de la réaction en chaîne par polymérase (PCR) a conduit au développement d'un grand nombre de variantes de la PCR.
Modifications de la méthode de base
[modifier | modifier le code]Souvent, seule une petite modification doit être apportée au protocole de la PCR standard pour atteindre l'objectif souhaité.
La PCR multiplexe utilise plusieurs paires d'amorces s'hybridant à différentes séquences cibles. Cela permet l'analyse simultanée de plusieurs cibles dans un seul échantillon. Par exemple, lors de tests de mutations génétiques, six amplifications ou plus peuvent être combinées. Dans le protocole standard pour les empreintes digitales d'ADN, les cibles testées sont souvent amplifiées en groupes de 3 ou 4. L'amplification de sonde dépendante de la ligature multiplex (ou MLPA) permet à plusieurs cibles d'être amplifiées en utilisant uniquement une seule paire d'amorces, ce qui évite les limitations de résolution de la PCR multiplexe. La PCR multiplexe a également été utilisée pour l'analyse des microsatellites et des SNP[1].

La PCR des nombres variables de répétitions en tandem (VNTR) cible les zones du génome qui présentent une longueur variable. L'analyse des génotypes de l'échantillon implique généralement le dimensionnement des produits d'amplification par électrophorèse sur gel. L'analyse de segments VNTR plus petits connus sous le nom de répétitions tandem courtes (ou STR) constitue le socle des bases de données d'empreintes digitales d'ADN telles que CODIS .
La PCR asymétrique amplifie préférentiellement un brin de l'ADN cible. Il est utilisé dans certaines méthodes de séquençage et chez les sondes d'hybridation, pour générer un brin d'ADN comme produit. Le thermocyclage est effectué comme en PCR classique, mais avec une quantité limitante d'amorces ou en en laissant une de côté. Lorsque l'amorce limitante devient épuisée, la réplication augmente de façon arithmétique par extension de l'amorce en excès[2]. Une modification de ce processus, nommée Linear-After-The-Exponential-PCR (ou LATE-PCR pour PCR tardive, litt.), utilise une amorce limitante ayant une température de fusion (Tm) plus élevée que l'amorce en excès, afin de maintenir l'efficacité de la réaction parce que la concentration d'amorce limitante diminue à mi-réaction[3]. (Voir également PCR de chevauchement-extension ou overlap-extension PCR en anglais.)
Certaines modifications sont nécessaires pour effectuer ce qu'on appelle une longue PCR. Le processus de PCR original basé sur fragments de Klenow n'a pas réussi à générer de produits supérieurs à environ 400 pb. La Taq polymérase peut cependant amplifier des cibles pouvant atteindre plusieurs milliers de pb (kb) de long[4]. Depuis lors, des protocoles modifiés avec l'enzyme Taq ont permis d'amplifier des cibles de plus de 50 kb[5].
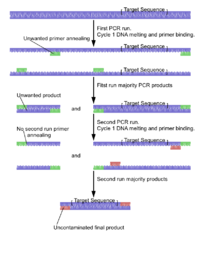
La PCR imbriquée est utilisée pour augmenter la spécificité de l'amplification de l'ADN. Deux jeux d'amorces sont utilisés dans deux réactions successives. Dans la première PCR, une paire d'amorces est utilisée pour générer des produits d'ADN, qui peuvent contenir des produits amplifiés à partir de zones non cibles. Les produits de la première PCR sont ensuite utilisés comme modèle dans une deuxième PCR, en utilisant une amorce ("hémi-imbrication") ou deux amorces différentes dont les sites de liaison sont localisés (imbriqués) dans le premier ensemble, augmentant ainsi la spécificité. La PCR imbriquée réussit souvent mieux à amplifier spécifiquement les produits d'ADN longs que la PCR conventionnelle, mais elle nécessite une connaissance plus détaillée de la séquence de la cible.
La PCR quantitative (ou qPCR) est utilisée pour mesurer la quantité spécifique d'ADN (ou ARN) cible dans un échantillon. En mesurant l'amplification uniquement dans la phase d'augmentation exponentielle réelle, la quantité de produit mesuré reflète plus précisément la quantité initiale de cible. Des thermocycleurs (machines à PCR) spéciaux sont utilisés pour surveiller la quantité de produit généré pendant l'amplification. Les méthodes de PCR quantitative en temps réel (qRT-PCR) utilisent des fluorochromes, tels que le SybrGreen, ou des sondes d'ADN contenant un fluorochrome, telles que TaqMan, pour mesurer la quantité de produit amplifié à mesure que la réaction progresse.
La PCR à démarrage à chaud (hot-start PCR) est une technique effectuée manuellement en chauffant les composants de la réaction à la température de fusion de l'ADN (par exemple 95 °C) avant d'ajouter la polymérase. De cette façon, on évite une amplification non spécifique à des températures plus basses[6]. Alternativement, des réactifs spécialisés inhibent l'activité de la polymérase à température ambiante, soit par la liaison d'un anticorps, soit par la présence d'inhibiteurs liés de façon covalente qui ne se dissocient qu'après une étape d'activation à haute température. La "PCR à démarrage à chaud/finition à froid" ("hot-start/cold-finish PCR") est obtenue avec de nouvelles polymérases hybrides qui sont inactives à température ambiante et ne sont activées qu'à des températures élevées.
Dans la PCR par essais (touchdown PCR), la température d'hybridation diminue progressivement au cours des cycles ultérieurs. La température d'hybridation dans les premiers cycles est généralement de 3 à 5 °C au-dessus de la norme Tm des amorces utilisées, tandis que dans les cycles ultérieurs, elle est de 3 à 5 °C en-dessous de la Tm. La température d'hybridation initiale plus élevée conduit à une plus grande spécificité pour la liaison des amorces, tandis que les températures plus basses permettent une amplification plus efficace à la fin de la réaction[7].
La PCR assemblée (assembly PCR, également connue sous le nom de Polymerase Cycling Assembly ou PCA) consiste à synthétiser de longues structures d'ADN en effectuant la PCR sur un pool d'oligonucléotides longs avec de courts segments se chevauchant, pour assembler deux ou plusieurs morceaux d'ADN en un seul morceau. Elle implique une PCR initiale avec des amorces qui se chevauchent et une seconde PCR utilisant les produits comme modèle permettant de générer le produit final de pleine longueur. Cette technique peut remplacer l'assemblage basé sur la ligature[8].
Dans la PCR coloniale (colony PCR), des colonies bactériennes sont criblées directement par PCR, par exemple lors du criblage pour obtenir des constructions de vecteurs d'ADN correctes. Les colonies sont échantillonnées avec une pointe de pipette stérile et une petite quantité de cellules est transférée dans un mélange pour PCR. Pour libérer l'ADN des cellules, la PCR est soit démarrée avec un temps prolongé à 95 °C (lorsque la polymérase standard est utilisée), ou avec une étape de dénaturation raccourcie à 100 °C et une ADN polymérase chimérique spéciale[9].
La PCR ditigale (digital PCR) permet d'amplifier simultanément des milliers d'échantillons, chacun isolé dans une gouttelette distincte au sein d'une émulsion.
La PCR suicide (suicide PCR) est généralement utilisée en paléogénétique ou dans d'autres études pour lesquelles éviter les faux positifs et garantir la spécificité du fragment amplifié est la plus haute priorité. Elle a été initialement décrite dans une étude visant à vérifier la présence de la bactérie Yersinia pestis dans des échantillons dentaires provenant de tombes du XIVe siècle de personnes qui auraient été tuées par la peste pendant l'épidémie médiévale de la peste noire[10]. La méthode prescrit l'utilisation de toute combinaison d'amorces une seule fois dans une PCR (d'où le terme "suicide"), qui n'aurait jamais dû être utilisée dans une réaction de PCR de contrôle positif, et les amorces doivent toujours cibler une région génomique jamais amplifiée auparavant dans le laboratoire utilisant ce ou tout autre ensemble d'amorces. Cela garantit qu'aucun ADN contaminant des réactions de PCR précédentes n'est présent dans le laboratoire, ce qui pourrait sinon générer des faux positifs.
La COLD-PCR (litt. "PCR à froid", acronyme de co-amplification at lower denaturation temperature-PCR, en français PCR de co-amplification à faible température de dénaturation) est un protocole modifié de PCR qui enrichit les variants alléliques d'un mélange de type sauvage ainsi que l'ADN contenant la mutation d'intérêt.
Prétraitements et extensions
[modifier | modifier le code]Le processus de base de la PCR peut parfois précéder ou suivre une autre technique de biologie moléculaire.
La RT-PCR (pour Reverse Transcription PCR ou PCR à transcriptase inverse en français) est utilisée pour la transcription inverse et l'amplification de l'ARN en ADNc. La PCR est précédée d'une réaction utilisant la transcriptase inverse, une enzyme issue des virus qui convertit l'ARN en ADNc. Les deux réactions peuvent être combinées dans un tube, l'étape de chauffage initiale de la PCR étant utilisée pour inactiver la transcriptase[4]. La polymérase Tth (décrite plus bas) a une activité rétrotranscriptase et peut effectuer toute la réaction. La RT-PCR est largement utilisée dans le profilage d'expression, qui permet de détecter le niveau d'expression d'un gène. Il peut également être utilisé pour obtenir la séquence d'un transcrit d'ARN, ce qui peut aider à la détermination des sites de début et de fin de transcription (par RACE-PCR) et faciliter la cartographie de l'emplacement des exons et des introns dans une séquence de gènes.
La PCR bilatérale (two-tailed PCR) utilise une seule amorce qui se lie à une cible de micro-ARN avec à la fois les extrémités 3' et 5', appelées hémi-sondes[11]. Les deux extrémités doivent être complémentaires pour que la liaison se produise. L'extrémité 3' est ensuite étendue par la transcriptase inverse, ce qui forme un long ADNc. L'ADNc est ensuite amplifié en utilisant deux amorces de PCR spécifiques à la cible. La combinaison de deux hémi-sondes, ciblant toutes deux la courte cible de micro-ARN, rend la PCR bilatérale extrêmement sensible et spécifique.
La PCR médiée par ligature (ligation-mediated PCR) utilise de petits "linkers" (ou adaptateurs) d'oligonucléotides d'ADN qui sont d'abord ligaturés à des fragments de l'ADN cible. Les amorces de PCR qui s'hybrident aux séquences de liaison sont ensuite utilisées pour amplifier les fragments cibles. Cette méthode est déployée pour le séquençage de l'ADN, l'arpentage chromosomique et le footprinting de l'ADN[12]. Une technique apparentée est le polymorphisme de longueur des fragments amplifiés (AFLP), qui génère des fragments de diagnostic d'un génome.
La PCR spécifique à la méthylation (MSP-PCR) est utilisée pour identifier les modèles de méthylation de l'ADN au niveau des îlots de cytosine-guanine (CpG) dans l'ADN génomique[13]. L'ADN cible est d'abord traité avec du bisulfite de sodium, qui convertit les bases de cytosine non méthylées en uracile, qui est complémentaire de l'adénosine dans les amorces de PCR. Deux amplifications sont ensuite effectuées sur l'ADN traité au bisulfite : un ensemble d'amorces est hybridé à l'ADN avec des cytosines (correspondant à la partie des cytosines méthylées), et l'autre ensemble est hybridé à l'ADN avec l'uracile (correspondant à la partie des cytosines non méthylées). La MSP utilisée en qPCR fournit des informations quantitatives sur l'état de méthylation d'un dinucléotide CpG donné[14].
Autres modifications
[modifier | modifier le code]Les ajustements dans les composants de la PCR sont couramment utilisés pour optimiser les performances.
L'ion magnésium divalent (Mg2+) est requis pour l'activité de la polymérase pendant la PCR. Des concentrations plus faibles en Mg2+ augmenteront la fidélité de réplication, tandis que des concentrations plus élevées introduiront plus de mutations[réf. nécessaire].
Les dénaturants (tels que le DMSO) peuvent augmenter la spécificité d'amplification en déstabilisant les liaisons d'amorces non spécifiques. D'autres produits chimiques, tels que le glycérol, sont des stabilisateurs de l'activité de la polymérase pendant l'amplification. Les détergents (comme le Triton X-100) peuvent empêcher la polymérase de coller à elle-même ou aux parois du tube de réaction.
Les ADN polymérases incorporent occasionnellement des bases de mésappariement dans le brin d'élongation. La PCR haute fidélité (high-fidelity PCR) utilise des enzymes avec une activité exonucléase 3'-5' qui diminue ce taux de mauvaise incorporation. Parmi les exemples d'enzymes ayant une activité de relecture, on peut citer la Pfu ; les ajustements des concentrations de Mg2+ et de dNTP peuvent aider à maximiser le nombre de produits qui correspondent exactement à l'ADN cible d'origine[réf. nécessaire].
Modifications des amorces
[modifier | modifier le code]Les ajustements aux oligonucléotides synthétiques utilisés comme amorces dans la PCR constituent une riche source de modification.
Normalement, les amorces de PCR sont choisies dans une partie invariante du génome et peuvent être utilisées pour amplifier une zone polymorphe entre elles. Dans la PCR spécifique aux allèles (allele-specific PCR), c'est le contraire qui se passe. Au moins une des amorces est choisie dans une zone polymorphe, avec les mutations situées à (ou près) de son extrémité 3'. Dans des conditions strictes, une amorce non appariée n'initiera pas la réplication, tandis qu'une amorce appariée le fera. L'apparition d'un produit d'amplification indique donc le génotype.
La PCR spécifique à InterSéquence (InterSequence-Specific PCR ou ISSR-PCR) est une méthode d'empreinte génétique qui utilise des amorces sélectionnées à partir de segments répétés tout au long d'un génome pour produire une empreinte digitale unique de longueurs de produit amplifiées[15]. L'utilisation d'amorces d'un segment fréquemment répété est appelée Alu-PCR et peut aider à amplifier les séquences adjacentes à (ou entre) ces répétitions.
Les amorces peuvent également être conçues pour être "dégénérées" - capables d'initier la réplication à partir d'un grand nombre d'emplacements cibles. L'amplification du génome entier (ou WGA pour Whole Genome Amplification) est un ensemble de procédures qui permettent à l'amplification de se produire à de nombreux endroits dans un génome inconnu, et qui ne peuvent être disponibles qu'en petites quantités. D'autres techniques utilisent des amorces dégénérées qui sont synthétisées en utilisant plusieurs nucléotides à des positions particulières (la polymérase «choisit» les amorces correctement appariées). En outre, les amorces peuvent être synthétisés avec l'analogue de nucléoside inosine, qui s'hybride à trois des quatre bases normales. Une technique similaire peut forcer la PCR à effectuer une mutagenèse dirigée vers le site-cible.
Normalement, les amorces utilisées dans la PCR sont conçues pour être pleinement complémentaires de la cible. Cependant, la polymérase est tolérante aux erreurs d'appariement en dehors de l'extrémité 3'. Les amorces à queue comprennent des séquences non complémentaires à leurs extrémités 5'. Une procédure courante est l'utilisation d'amorces de liaison, qui placent finalement des sites de restriction aux extrémités des produits de PCR, facilitant leur insertion ultérieure dans des vecteurs de clonage.
Une extension de la méthode "colony-PCR" (décrite plus haut) est l'utilisation d'amorces vectorielles. Les fragments d'ADN cible (ou d'ADNc) sont d'abord insérés dans un vecteur de clonage, et un seul ensemble d'amorces est conçu pour les zones du vecteur flanquant le site d'insertion. L'amplification se produit pour tout ADN inséré[4].
La PCR peut facilement être modifiée pour générer un produit marqué destiné à une utilisation ultérieure comme sonde d'hybridation. Une ou les deux amorces peuvent être utilisées en PCR avec un marqueur radioactif ou fluorescent déjà attaché, ou bien des marqueurs peuvent être ajoutés après l'amplification. Ces méthodes de marquage peuvent être combinées avec une "PCR asymétrique" (voir plus haut) pour produire des sondes d'hybridation efficaces.
La PCR dépendante de la RNase H (rhPCR) peut réduire la formation d'amorce-dimère et augmenter le nombre d'analyses dans la PCR multiplexe. Le procédé utilise des amorces avec un bloc clivable à l'extrémité 3' qui est éliminé par l'action d'une enzyme RNase HII thermostable[16].
ADN polymérases
[modifier | modifier le code]Il existe plusieurs ADN polymérases qui sont utilisées dans la PCR.
Le fragment de Klenow, dérivé de l'ADN polymérase I provenant d'E. coli, a été la première enzyme utilisée en PCR. En raison de son manque de stabilité à haute température, elle doit être réapprovisionnée au cours de chaque cycle et n'est donc pas couramment utilisé en PCR.
L'ADN polymérase du bactériophage T4 (famille A) a également été initialement utilisée en PCR. Elle a une fidélité de réplication plus élevée que le fragment de Klenow, mais est également détruite par la chaleur. L'ADN polymérase T7 (famille B) a des propriétés et des objectifs similaires. Elle a été appliquée à la mutagenèse dirigée[17] et au séquençage de Sanger[18].
La polymérase Taq, l'ADN polymérase I de la bactérie Thermus aquaticus, a été la première polymérase thermostable utilisée en PCR et est toujours la plus utilisée. L'enzyme peut être isolée de sa source native ou de son gène cloné exprimé dans E. coli[4]. Un fragment de 61 kDa, dépourvu d'activité exonucléase 5'-3', est connu sous le nom de fragment de Stoffel et est exprimé chez E. coli[19]. Le manque d'activité exonucléase peut lui permettre d'amplifier des cibles plus longues que l'enzyme native. Il a été commercialisé sous les noms AmpliTaq et Klentaq[20]. Une variante conçue pour la PCR à démarrage à chaud appelée "polymérase Faststart" a également été produite. Elle nécessite une forte activation thermique, évitant ainsi une amplification non spécifique due à l'activité de la polymérase à basse température. De nombreuses autres variantes ont été créées[21].
D'autres polymérases issues du genre Thermus, telles que la polymérase Tth I (référence UniProt : P52028) provenant de Thermus thermophilus, ont été utilisées. Tth a une activité de transcriptase inverse en présence d'ions Mn2+, permettant une amplification par PCR à partir de cibles d'ARN[22].
Le genre archéen Pyrococcus s'est révélé une riche source de polymérases thermostables avec une activité de correction sur épreuves. L'ADN polymérase Pfu, isolée de P. furiosus, montre une diminution de 5 fois du taux d'erreur de réplication par rapport à la Taq[23]. Étant donné que les erreurs augmentent à mesure que la PCR progresse, Pfu est la polymérase que l'on préférera lorsque les produits doivent être clonés individuellement pour le séquençage ou l'expression. D'autres polymérases moins utilisées de ce genre comprennent la Pwo (UniProt : P61876) provenant de Pyrococcus woesei, la Pfx d'une espèce sans nom, ou encore la polymérase "Deep Vent" (UniProt : Q51334) issue de la souche GB-D[24].
La polymérase Vent ou Tli est une ADN polymérase extrêmement thermostable isolée de Thermococcus litoralis. La polymérase de Thermococcus fumicolans (Tfu) a également été commercialisée[24].
Modifications du mécanisme
[modifier | modifier le code]Parfois, même le mécanisme de base de la PCR peut être modifié.
Contrairement à la PCR normale, la PCR inverse permet l'amplification et le séquençage de l'ADN qui entoure une séquence connue. Il s'agit d'abord de soumettre l'ADN cible à une série de digestions par des enzymes de restriction, puis de circulariser les fragments résultants par auto-ligation. Les amorces sont conçues pour être étendues vers l'extérieur à partir du segment connu, ce qui entraîne une amplification du reste du cercle. Ceci est particulièrement utile pour identifier des séquences de chaque côté de divers inserts génomiques[25].
De même, la PCR entrelacée thermique asymétrique (ou TAIL-PCR pour thermal asymmetric interlaced PCR) est utilisée pour isoler des séquences inconnues flanquant une zone connue du génome. Dans la séquence connue, la TAIL-PCR utilise une paire d'amorces imbriquées avec des températures d'hybridation différentes. Une amorce "dégénérée" est utilisée pour amplifier dans l'autre sens à partir de la séquence inconnue[26].
Méthodes d'amplification isotherme
[modifier | modifier le code]Certains protocoles d'amplification d'ADN ont été développés et peuvent être utilisés alternativement à la PCR. Ils sont isothermes, ce qui signifie qu'ils fonctionnent à température constante.
L'amplification dépendante de l'hélicase est similaire à la PCR traditionnelle, mais utilise une température constante plutôt que de parcourir les étapes classiques de dénaturation et d'hybridation/extension. L'hélicase, une enzyme qui déroule l'ADN, est utilisée à la place de la dénaturation thermique[27]. L'amplification isotherme médiée par les boucles est une idée similaire, mais réalisée avec une polymérase à déplacement de brin[28].
La réaction d'amplification enzymatique de coupure (NEAR pour nicking enzyme amplification reaction) est isotherme, produisant des copies d'ADN à une température constante à l'aide d'une polymérase et d'une enzyme de coupure.
L'amplification de la polymérase de la recombinase (RPA pour recombinase polymerase amplification)[29] utilise une recombinase pour coupler spécifiquement des amorces avec de l'ADN double brin sur la base de leur homologie, dirigeant ainsi la synthèse d'ADN à partir de séquences d'ADN définies présentes dans l'échantillon. La présence de la séquence cible initie l'amplification de l'ADN et aucune fusion thermique ou chimique de l'ADN n'est requise. La réaction progresse rapidement et se traduit par une amplification spécifique de l'ADN à partir de quelques copies cibles à des niveaux détectables généralement en 5 à 10 minutes. L'ensemble du système réactionnel est stable sous forme de formulation séchée et n'a pas besoin d'être réfrigéré. La RPA peut être utilisée pour remplacer la PCR dans diverses applications de laboratoire et les utilisateurs peuvent concevoir leurs propres tests[30].
Voir aussi
[modifier | modifier le code]Notes
[modifier | modifier le code]- (en) Cet article est partiellement ou en totalité issu de l’article de Wikipédia en anglais intitulé « Variants of PCR » (voir la liste des auteurs).
Références
[modifier | modifier le code]- ↑ « Multiplex-Ready PCR: A new method for multiplexed SSR and SNP genotyping », BMC Genomics, vol. 9, , p. 80 (PMID 18282271, PMCID 2275739, DOI 10.1186/1471-2164-9-80)
- ↑ « DNA sequencing with Thermus aquaticus DNA polymerase and direct sequencing of polymerase chain reaction-amplified DNA », Proc. Natl. Acad. Sci. U.S.A., vol. 85, no 24, , p. 9436–40 (PMID 3200828, PMCID 282767, DOI 10.1073/pnas.85.24.9436, Bibcode 1988PNAS...85.9436I)
- ↑ Linear-after-the-exponential polymerase chain reaction and allied technologies Real-time detection strategies for rapid, reliable diagnosis from single cells, vol. 132, coll. « Methods in Molecular Medicine™ », , 65–85 p. (ISBN 978-1-58829-578-1, PMID 17876077, DOI 10.1007/978-1-59745-298-4_7)
- Saiki RK, Gelfand DH, Stoffel S et Scharf, « Primer-directed enzymatic amplification of DNA with a thermostable DNA polymerase », Science, vol. 239, no 4839, , p. 487–91 (PMID 2448875, DOI 10.1126/science.239.4839.487, Bibcode 1988Sci...239..487S, lire en ligne)
- ↑ « Effective amplification of long targets from cloned inserts and human genomic DNA », Proc. Natl. Acad. Sci. U.S.A., vol. 91, no 12, , p. 5695–9 (PMID 8202550, PMCID 44063, DOI 10.1073/pnas.91.12.5695, Bibcode 1994PNAS...91.5695C)
- ↑ « Prevention of pre-PCR mis-priming and primer dimerization improves low-copy-number amplifications », Nucleic Acids Res., vol. 20, no 7, , p. 1717–23 (PMID 1579465, PMCID 312262, DOI 10.1093/nar/20.7.1717, lire en ligne)
- ↑ « 'Touchdown' PCR to circumvent spurious priming during gene amplification », Nucleic Acids Res., vol. 19, no 14, , p. 4008 (PMID 1861999, PMCID 328507, DOI 10.1093/nar/19.14.4008, lire en ligne)
- ↑ « Single-step assembly of a gene and entire plasmid from large numbers of oligodeoxyribonucleotides », Gene, vol. 164, no 1, , p. 49–53 (PMID 7590320, DOI 10.1016/0378-1119(95)00511-4)
- ↑ DNA Sequencing II : Optimizing Preparation and Cleanup, Jones and Bartlett, , 241–257 p. (ISBN 978-0-7637-3383-4, lire en ligne), « Thermostable DNA Polymerases for a Wide Spectrum of Applications: Comparison of a Robust Hybrid TopoTaq to other enzymes »
- ↑ Raoult, G Aboudharam, E Crubezy et G Larrouy, « Molecular identification by "suicide PCR" of Yersinia pestis as the agent of medieval black death », Proc. Natl. Acad. Sci. U.S.A., vol. 97, no 23, , p. 12800–12803 (ISSN 0027-8424, PMID 11058154, PMCID 18844, DOI 10.1073/pnas.220225197, Bibcode 2000PNAS...9712800R)
- ↑ Androvic, Valihrach, Elling et Sjoback, « Two-tailed RT-qPCR: a novel method for highly accurate miRNA quantification », Nucleic Acids Research, vol. 45, no 15, , e144 (ISSN 0305-1048, PMID 28911110, PMCID 5587787, DOI 10.1093/nar/gkx588)
- ↑ « In vivo footprinting of a muscle specific enhancer by ligation mediated PCR », Science, vol. 246, no 4931, , p. 780–6 (PMID 2814500, DOI 10.1126/science.2814500, Bibcode 1989Sci...246..780M)
- ↑ « Methylation-specific PCR: a novel PCR assay for methylation status of CpG islands », Proc. Natl. Acad. Sci. U.S.A., vol. 93, no 18, , p. 9821–6 (PMID 8790415, PMCID 38513, DOI 10.1073/pnas.93.18.9821, Bibcode 1996PNAS...93.9821H)
- ↑ Hernández, Tse, Pang et Arboleda, « Optimizing methodologies for PCR-based DNA methylation analysis. », BioTechniques, vol. 55, no 4, , p. 181–197 (PMID 24107250, DOI 10.2144/000114087)
- ↑ E. Zietkiewicz, A. Rafalski et D. Labuda, « Genome fingerprinting by simple sequence repeat (SSR)-anchored polymerase chain reaction amplification », Genomics, vol. 20, no 2, , p. 176–83 (PMID 8020964, DOI 10.1006/geno.1994.1151)
- ↑ « RNase H-dependent PCR (rhPCR): improved specificity and single nucleotide polymorphism detection using blocked cleavable primers », BMC Biotechnology, vol. 11, , p. 80 (PMID 21831278, PMCID 3224242, DOI 10.1186/1472-6750-11-80)
- ↑ « Use of modified T7 DNA polymerase (sequenase version 2.0) for oligonucleotide site-directed mutagenesis », Nucleic Acids Research, vol. 17, no 8, , p. 3314 (PMID 2726477, PMCID 317753, DOI 10.1093/nar/17.8.3314)
- ↑ « Thermo Sequenase DNA Polymerase »
- ↑ Lawyer, Stoffel, Saiki et Chang, « High-level expression, purification, and enzymatic characterization of full-length Thermus aquaticus DNA polymerase and a truncated form deficient in 5' to 3' exonuclease activity », PCR Methods and Applications, vol. 2, no 4, , p. 275–287 (ISSN 1054-9803, PMID 8324500, DOI 10.1101/gr.2.4.275)
- ↑ « Applied Biosystems - Support », www6.appliedbiosystems.com
- ↑ Villbrandt, Sobek, Frey et Schomburg, « Domain exchange: chimeras of Thermus aquaticus DNA polymerase, Escherichia coli DNA polymerase I and Thermotoga neapolitana DNA polymerase. », Protein Engineering, vol. 13, no 9, , p. 645–54 (PMID 11054459, DOI 10.1093/protein/13.9.645, lire en ligne)
- ↑ https://www.promega.in/products/pcr/rt-pcr/tth-dna-polymerase/
- ↑ « PCR fidelity of pfu DNA polymerase and other thermostable DNA polymerases », Nucleic Acids Res., vol. 24, no 18, , p. 3546–51 (PMID 8836181, PMCID 146123, DOI 10.1093/nar/24.18.3546, lire en ligne)
- Elizabeth van Pelt-Verkuil, Alex van Belkum et John P. Hays, Principles and Technical Aspects of PCR Amplification, , 103–18 p. (ISBN 978-1-4020-6240-7, DOI 10.1007/978-1-4020-6241-4_7), « Taq and Other Thermostable DNA Polymerases »
- ↑ « Genetic Applications of an Inverse Polymerase Chain Reaction », Genetics, vol. 120, no 3, , p. 621–3 (PMID 2852134, PMCID 1203539, lire en ligne)
- ↑ « Thermal asymmetric interlaced PCR: automatable amplification and sequencing of insert end fragments from P1 and YAC clones for chromosome walking », Genomics, vol. 25, no 3, , p. 674–81 (PMID 7759102, DOI 10.1016/0888-7543(95)80010-J)
- ↑ « Helicase-dependent isothermal DNA amplification », EMBO Rep., vol. 5, no 8, , p. 795–800 (PMID 15247927, PMCID 1249482, DOI 10.1038/sj.embor.7400200)
- ↑ « Loop-mediated isothermal amplification of DNA », Nucleic Acids Res., vol. 28, no 12, , p. 63e–63 (PMID 10871386, PMCID 102748, DOI 10.1093/nar/28.12.e63)
- ↑ « DNA Detection Using Recombination Proteins », PLOS Biol, vol. 4, no 7, , e204 (PMID 16756388, PMCID 1475771, DOI 10.1371/journal.pbio.0040204)
- ↑ « Microfluidic lab-on-a-foil for nucleic acid analysis based on isothermal recombinase polymerase amplification (RPA) », Lab Chip, vol. 10, no 7, , p. 887–93 (PMID 20300675, DOI 10.1039/b921140c)


 French
French Deutsch
Deutsch