DNA fetale libero circolante
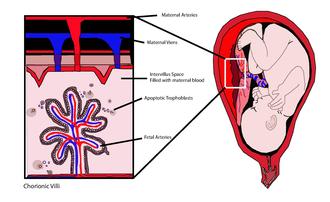
Per DNA fetale libero circolante (in inglese Cell-free fetal DNA o cffDNA) si intende il DNA del feto che si trova a circolare liberamente nel flusso sanguigno materno. Esso può essere raccolto tramite un prelievo venoso sulla madre. L'analisi di questo DNA può fornire un metodo di screening, ma non di diagnosi prenatale non invasiva.
Il DNA fetale libero circolante proviene dal trofoblasto (un tessuto che fornisce nutrimento all'embrione) che forma la placenta.[1][2] Si stima che il 2-6% del DNA nel sangue materno ha origine fetale.[3]
Già nel 1983 era stata dimostrata la presenza con cariotipo maschile nel sangue di donne in gravidanza con feto maschio. In questo studio venne utilizzata la tecnica di Polymerase chain reactin (PCR) per avere la conferma della presenza di queste cellule fetali nel circolo materno. L’analisi fu condotta su un campione di 43 donne con gestazione compresa tra le 12 e le 40 settimane. Trenta di queste gravide avevano un feto maschio, mentre 13 erano le gravide con feto femmina. Il risultato dello studio fu che le cellule con cromosoma Y vennero riscontrate solo nelle donne con feto maschio. Il DNA libero circolante nel sangue materno è una commistione di DNA materno e DNA placentare derivato dall’apoptosi delle cellule del citotrofoblasto, in particolare del sinciziotrofoblasto che compone i tessuti placentari,
il DNA fetale libero scompare dopo poco tempo dal termine della gavidanza, mentre le cellule fetali integre possono sopravvivre per più tempo. Il fatto di avere una rapida clearence rende il cffDNA (cell fetal free DNA) gravidanza specifico[4].
Il DNA fetale è frammentato e si fa strada nel sangue materno tramite spargimento delle microparticelle placentari nella circolazione sanguigna materna.[5] Alcuni studi hanno dimostrato che il DNA fetale può essere trovato già alla 7ª settimana di gestazione e la sua quantità aumenta con il progredire della gravidanza[6], per poi diminuire rapidamente dopo la nascita del bambino, arrivando a non essere più rilevabile nel sangue materno circa 2 ore dopo la nascita.[7] Il DNA fetale libero circolante è significativamente più piccolo del DNA materno, con frammenti di circa 200pb.[8] Molti metodi utili ad estrarre il DNA fetale dal plasma materno utilizzano le sue dimensioni per distinguerlo da quello della madre.[9][10]
Può essere utilizzato nell'ambito della diagnosi prenatale a partire dalla 10ª settimana di gestazione quando cioè la quantità di DNA presente è sufficiente per condurre analisi[4].
Il valore del cffDNA è espresso da una percentuale definita frazione fetale (FF). il valore della FF varia con il progredire della gravidanza, può essere influenzato da molteplici fattori quali ad esempio l’indice di massa corporea materno, in quanto, per un fenomeno sovrapponibile alla diluizione, la FF diminuisce con l’aumentare del BMI. La FF aumenta con il progredire della gravidanza in quanto aumentano le dimensioni della placenta e di conseguenza il numero di cellule che vanno incontro ad apoptosi del trofoblasto. Nelle gravidanze plurime il valore della FF è aumentato rispetto alle gravidanze singole, anche in questo caso si ritiene che tale fenomeno possa essere legato alle maggiori dimensioni della placenta (se unica) o alla presenza di due placente, con la conseguenza che le cellule trofoblastiche che vanno incontro all’apoptosi è maggiore rispetto alle gravidanze singole[4].
Anche le alterazioni cromosomiche sono in grado di alterare la FF. Questa risulta essere più alta nei feti con trisomia 21. Al contrario si osservano livelli più bassi di FF in feti affetti da trisomia 13, 18, o monosomia del cromosoma X.
Per l’analisi del cffDNA nell’ambito della diagnosi prenatale, la FF deve avere un valore superiore al 4%. In talune donne il valore della FF non supera questo cut off, in questi casi può essere ripetuto il prelievo di sangue materno nella speranza che il proseguimento della gravidanza abbia aumentato il livello della FF[4].
In caso il criterio di sufficienza della FF sia soddisfatto si passa all’ analisi delle aneuploidie tramite NIPT. Il NIPT è un test di screening e non un test diagnostico proprio per il fatto che le tecniche di analisi non valutano solo il cffDNA ma il cfDNA (cell freeDNA) totale includendo sia il DNA libero circolante materno che fetale. La sensibilità e specificità del NIPT sono maggiori rispetto al test combinato per l’analisi delle trisomie 13,18,21[4].
Il risultato possibile per l’esame NIPT è di alto o basso rischio per una specifica trisomia, ad esempio alto rischio per trisomia 13[4].
In caso di risultato di alto rischio per un’aneuploidia è necessario procedere all’esecuzione della diagnosi invasiva[4].
Gli studi hanno esaminato, e in alcuni casi anche ottimizzato, i protocolli per la determinazione dei fattori RhD non compatibili, per la determinazione delle malattie genetiche legate ai cromosomi sessuali e per le malattie ereditarie.[11] Gli studi in corso stanno ora cercando di determinare le aneuploidie nel feto in via di sviluppo.[12][13] Questi test possono essere condotti più precocemente rispetto agli attuali metodi diagnostici prenatali e non presentano alcun rischio di aborto spontaneo.[14] Il test prenatale non invasivo (NIPT), questo è il termine oggi usato invece di diagnosi prenatale non invasiva (NIPD), è utilizzato anche in Europa e in parte degli Stati Uniti e come metodica è in continuo sviluppo; è quasi certo che in un prossimo futuro ridurranno il numero di casi considerati a rischio e per i quali rimane necessario ricorrere a prelievo di villi coriali (CVS) e l'amniocentesi, che sono tuttora le uniche metodiche di diagnosi.[15], che hanno rischi di aborto spontaneo correlati alla procedura rispettivamente di circa 1 su 100 gravidanze e 1 su 200.[16][17]
Come metodo di diagnosi prenatale, le tecniche basate sul DNA fetale libero circolante condividono gli stessi problemi etici e pratici.[11]
Note
[modifica | modifica wikitesto]- ^ Alberry, M., et al. (2007). "Free fetal DNA in maternal plasma in anembryonic pregnancies: confirmation that the origin is the trophoblast". Prenatal Diagnosis 27 (5): 415-418.
- ^ Gupta, A. K., et al. (2004). "Detection of fetal DNA and RNA in placenta-derived syncytiotrophoblast microparticles generated in vitro". Clinical Chemistry 50 (11): 2187-2190.
- ^ Lo, E. S.; Lo, Y. M.; Hjelm, N. M.; Thilaganathan, B. (1998). "Transfer of nucleated maternal cells into fetal circulation during the second trimester of pregnancy". British Journal of Haematology 100 (3): 605-606.
- ^ a b c d e f g Manuale di Genetica per le professioni sanitarie. Autore Marco Tanini, Elform e-learning SRL Editore. Latina 2022, ISBN 978-88909408-0-4.
- ^ Smets, E. M.; Visser, A.; Go, A. T.; van Vugt, J. M.; Oudejans, C. B. (2006). "Novel biomarkers in preeclampsia". Clinica Chimica Acta: International Journal of Clinical Chemistry 364 (1-2): 22-32.
- ^ Lo, Y. M., et al. (1998). "Quantitative analysis of fetal DNA in maternal plasma and serum: implications for noninvasive prenatal diagnosis". American Journal of Human Genetics. 62 (4): 768-775.
- ^ Lo, Y. M., et al. (1999). "Rapid clearance of fetal DNA from maternal plasma". American Journal of Human Genetics 64 (1): 218-224.
- ^ Chan, K. C., et al. (2004). "Size distributions of maternal and fetal DNA in maternal plasma". Clinical Chemistry 50 (1): 88-92.
- ^ Li, Y., et al. (2004). "Size separation of circulatory DNA in maternal plasma permits ready detection of fetal DNA polymorphisms". Clinical Chemistry 50 (6): 1002-1011.
- ^ Li, Y., et al. (2005). "Detection of paternally inherited fetal point mutations for beta-thalassemia using size-fractionated cell-free DNA in maternal plasma". The Journal of the American Medical Association 293 (7): 843-849.
- ^ a b Hahn, S.; Chitty, L. S. (2008). "Noninvasive prenatal diagnosis: current practice and future perspectives". Current Opinion in Obstetrics & Gynecology 20 (2): 146-151.
- ^ Fan, H. C.; Blumenfeld, Y. J.; Chitkara, U.; Hudgins, L.; Quake, S. R. (2008). "Noninvasive diagnosis of fetal aneuploidy by shotgun sequencing DNA from maternal blood". Proceedings of the National Academy of Sciences of the United States of America 105 (42): 16266-16271.
- ^ Lo, Y. M., et al. (2007). "Digital PCR for the molecular detection of fetal chromosomal aneuploidy". Proceedings of the National Academy of Sciences of the United States of America 104 (32): 13116-13121.
- ^ Allyse, M.; Sayres, L. C.; King, J. S.; Norton, M. E.; Cho, M. K. (2012). "Cell-free fetal DNA testing for fetal aneuploidy and beyond: clinical integration challenges in the US context". Human Reproduction 27 (11): 3123-3131.
- ^ Lo, Y. M., et al. (1998). "Prenatal diagnosis of fetal RhD status by molecular analysis of maternal plasma". The New England Journal of Medicine 339 (24): 1734-1738.
- ^ Mujezinovic, F.; Alfirevic, Z. (2007). "Procedure-related complications of amniocentesis and chorionic villous sampling: a systematic review". Obstetrics and Gynecology 110 (3): 687-694.
- ^ Lo, Y. M. (2008). "Fetal nucleic acids in maternal plasma". Annals of the New York Academy of Sciences 1137: 140-143.
First trimester contingent screening for trisomy 21 by biomarkers and maternal blood cell-free DNA test - K.H. Nicolaides et others Ultrasound obstet Gynecol 2013;42:41-50
Non si può parlare di NIPD che comporta implicito il concetto di diagnosi,in quanto il test su DNA fetale nel sangue materno è un esame di screening, non potendo escludere la presenza di anomalie cromosomiche, ma calcolandone solo il rischio. E' corretto quindi utilizzare la sigla NIPT, cioè test di screening sul DNA circolante fetale.In nessun caso un tale test può sostituire il processo diagnostico basato su villocentesi o amniocentesi, che porta all'analisi del cariotipo fetale e alla diagnosi certa di anomalie cromosomiche o alla loro certa esclusione. Attualmente il NIPT è solo un calcolo probabilistico che ha costi troppo elevati per poter essere applicato a tutta la popolazione


 French
French Deutsch
Deutsch