Fluoreszenzmarkierung – Wikipedia
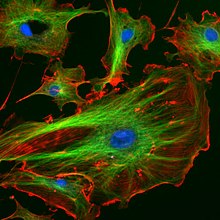
Die Fluoreszenzmarkierung ist eine Methode der Biochemie zur Markierung von Biomolekülen mit Fluorophoren.
Prinzip
[Bearbeiten | Quelltext bearbeiten]
Biomoleküle werden gelegentlich zur erleichterten Verfolgung mit fluoreszenten Molekülen versehen.[2] Durch die Kupplung von Fluorophoren an Proteine oder Kohlenhydrate auf der Zelloberfläche können auch Zellen direkt markiert werden oder mit fluoreszenten Immunkonjugaten indirekt nachgewiesen werden,[3] z. B. per Durchflusszytometrie, Fluoreszenzmikroskopie oder Fluoreszenztomographie.
Bei einer Fluoreszenzmarkierung kann das nachzuweisende Molekül direkt mit einem Fluorophor versehen werden oder indirekt durch selektiv bindende fluoreszente Moleküle nachgewiesen werden. Methoden zur Fluoreszenzmarkierung sind:
- Direkte Molekülnachweise
- chemische Kupplung (nur in vitro)
- bioorthogonale Markierung
- fluoreszente Reportermoleküle
- fluoreszente Protein-Tags, Protein-Tags mit Fluorophorkupplung (Flash-Tag) bzw. RNA-Tags
- Indirekte Molekülnachweise
Proteine
[Bearbeiten | Quelltext bearbeiten]
Bei Proteinen werden in vivo unter anderem das grün fluoreszierende Protein (GFP) als rekombinantes Fusionsprotein oder als Reportergen verwendet.[4] Inteine können zur C-terminalen Markierung von Proteinen eingesetzt werden. Als fluoreszentes Protein-Tag kann auch das Flash-Tag verwendet werden. Mit Redox-abhängigen Fluoreszenten Proteinen wie roGFP, rxYFP und HyPer kann die Fluoreszenz sauerstoffabhängig verändert werden. Mit spannungsabhängigen Proteinen wie VSFP kann die Fluoreszenz durch Anlegen einer elektrischen Spannung verändert werden.


Proteine können auch teilsynthetisch (bioorthogonal) markiert werden, in dem reaktive Aminosäurederivate in vivo eingebaut werden, die im Anschluss in vitro mit einem Fluorophor gekuppelt werden. Weiterhin werden Proteine mit verschiedenen chemischen Kupplungsreaktionen in vitro mit unterschiedlichen Fluorophoren versehen. Beispielsweise können nukleophile Gruppen (am häufigsten kommen Aminogruppen in Proteinen vor) durch Reaktionen mit Isothiocyanate (z. B. FITC, TRITC), Succinimidylestern (z. B. CFSE) mit unterschiedlichen Fluoreszenzfarbstoffen wie z. B. Fluorescein, Rhodamin und deren Derivate markiert werden. 1-Fluor-2,4-dinitrobenzol und Dansylchlorid reagieren mit Aminogruppen. Cysteine können mit IAEDANS oder Ellmans Reagenz markiert werden. Proteine können auch indirekt durch eine Immunmarkierung mit fluoreszenten Immunkonjugaten nachgewiesen werden, z. B. bei der Fluoreszenzmikroskopie, dem Western Blot und dem Immunoassay. Im Zuge einer Peptidsynthese können Peptide durch Verwendung von fluoreszenten Aminosäurederivaten markiert werden. Durch einen Förster-Resonanzenergietransfer (FRET), Biolumineszenz-Resonanzenergietransfer (BRET), Fluoreszenzkorrelationsspektroskopie (FCS) oder eine Bimolekulare Fluoreszenzkomplementation (BiFC) kann mit Fluorophoren die Nachbarschaft zweier Proteine nachgewiesen werden.
Nukleinsäuren
[Bearbeiten | Quelltext bearbeiten]DNA kann bioorthogonal markiert werden. Dazu kann DNA in vivo oder in vitro mit Nukleosidanaloga markiert werden, welche z. B. anschließend per Staudinger-Reaktion mit einem Fluorophor gekuppelt werden können.[5] DNA kann chemisch mit Fluorophoren versehen werden.[6] Oligonukleotide können durch eine Phosphoramidit-Synthese mit Fluorophoren markiert werden, die z. B. in der QPCR, der DNA-Sequenzierung und der In-situ-Hybridisierung verwendet werden. Daneben kann DNA enzymatisch im Zuge einer Polymerasekettenreaktion mit fluoreszenten Nukleotiden erzeugt oder mit einer Ligase oder einer terminalen Desoxynukleotidyltransferase markiert werden.[7][8][9] DNA kann indirekt durch eine Biotinylierung und fluoreszentem Avidin nachgewiesen werden. Als Fluorophore werden für Kupplungen unter anderem Fluorescein, fluoreszente Lanthanoide,[10][11] Gold-Nanopartikel, Kohlenstoffnanoröhren oder Quantenpunkte eingesetzt.[6]
Literatur
[Bearbeiten | Quelltext bearbeiten]- F. Borek: The fluorescent antibody method in medical and biological research. In: Bulletin of the World Health Organization. Band 24, Nummer 2, 1961, S. 249–256, PMID 20604086, PMC 2555496 (freier Volltext).
Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ S. J. Smith: Circuit reconstruction tools today. In: Current opinion in neurobiology. Band 17, Nummer 5, Oktober 2007, S. 601–608, doi:10.1016/j.conb.2007.11.004. PMID 18082394. PMC 2693015 (freier Volltext).
- ↑ C. P. Toseland: Fluorescent labeling and modification of proteins. In: Journal of chemical biology. Band 6, Nummer 3, April 2013, S. 85–95, doi:10.1007/s12154-013-0094-5. PMID 24432126. PMC 3691395 (freier Volltext).
- ↑ S. Lindström: Flow cytometry and microscopy as means of studying single cells: a short introductional overview. In: Methods in Molecular Biology. Band 853, 2012, S. 13–15, doi:10.1007/978-1-61779-567-1_2. PMID 22323136.
- ↑ D. Jung, K. Min, J. Jung, W. Jang, Y. Kwon: Chemical biology-based approaches on fluorescent labeling of proteins in live cells. In: Molecular bioSystems. Band 9, Nummer 5, Mai 2013, S. 862–872, doi:10.1039/c2mb25422k. PMID 23318293.
- ↑ C. C. Wang, T. S. Seo, Z. Li, H. Ruparel, J. Ju: Site-specific fluorescent labeling of DNA using Staudinger ligation. In: Bioconjugate Chemistry. Band 14, Nummer 3, 2003 May-Jun, S. 697–701, doi:10.1021/bc0256392. PMID 12757398.
- ↑ a b L. J. Kricka, P. Fortina: Analytical ancestry: "firsts" in fluorescent labeling of nucleosides, nucleotides, and nucleic acids. In: Clinical chemistry. Band 55, Nummer 4, April 2009, S. 670–683, doi:10.1373/clinchem.2008.116152. PMID 19233914.
- ↑ J. M. Prober, G. L. Trainor, R. J. Dam, F. W. Hobbs, C. W. Robertson, R. J. Zagursky, A. J. Cocuzza, M. A. Jensen, K. Baumeister: A system for rapid DNA sequencing with fluorescent chain-terminating dideoxynucleotides. In: Science (1987), Band 238(4825), S. 336–41. PMID 2443975.
- ↑ R. W. Richardson, R. I. Gumport: Biotin and fluorescent labeling of RNA using T4 RNA ligase. In: Nucleic acids research. Band 11, Nummer 18, September 1983, S. 6167–6184, PMID 6194506. PMC 326365 (freier Volltext).
- ↑ H. Eshaghpour, D. Söll, D. M. Crothers: Specific chemical labeling of DNA fragments. In: Nucleic acids research. Band 7, Nummer 6, November 1979, S. 1485–1495, PMID 228251. PMC 342322 (freier Volltext).
- ↑ J. Nurmi, T. Wikman, M. Karp, T. Lövgren: High-performance real-time quantitative RT-PCR using lanthanide probes and a dual-temperature hybridization assay. In: Analytical chemistry. Band 74, Nummer 14, Juli 2002, S. 3525–3532, PMID 12139064.
- ↑ A. Lehmusvuori, A. H. Tapio, P. Mäki-Teeri, K. Rantakokko-Jalava, Q. Wang, H. Takalo, T. Soukka: Homogeneous duplex polymerase chain reaction assay using switchable lanthanide fluorescence probes. In: Analytical biochemistry. Band 436, Nummer 1, Mai 2013, S. 16–21, doi:10.1016/j.ab.2013.01.007. PMID 23353013.


 French
French Deutsch
Deutsch