5-Méthylcytosine — Wikipédia
| 5-Méthylcytosine | ||
 | ||
 | ||
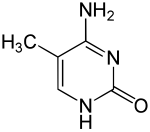
| Structure de la 5-méthylcytosine | ||
| Identification | ||
|---|---|---|
| Nom UICPA | 4-amino-5-méthylpyrimidin-2(1H)-one | |
| Synonymes | 5-méthylcytosine | |
| No CAS | ||
| No ECHA | 100.008.236 | |
| PubChem | 65040 | |
| ChEBI | 27551 | |
| SMILES | ||
| InChI | ||
| Propriétés chimiques | ||
| Formule | C5H7N3O [Isomères] | |
| Masse molaire[1] | 125,128 6 ± 0,005 4 g/mol C 47,99 %, H 5,64 %, N 33,58 %, O 12,79 %, | |
| Unités du SI et CNTP, sauf indication contraire. | ||
| modifier | ||
La 5-méthylcytosine est une base nucléique dérivée de la cytosine par méthylation sur l'atome de carbone no 5 du cycle pyrimidine. Cela modifie sa structure sans pour autant altérer ses propriétés d'appariement avec la guanine. Son nucléoside correspondant est la 5-méthylcytidine. Elle est impliquée dans la régulation de la transcription des gènes : lorsque la cytosine est méthylée dans l'ADN, la séquence de ce dernier n'est pas modifiée, mais l'expression de tels gènes méthylés peut être altérée ; l'étude de ce phénomène entre dans le cadre de l'épigénétique.
La 5-méthylcytosine est formée in vivo sous l'action d'une ADN méthyltransférase[2]. Sa distribution biologique dépend des espèces considérées :
- chez les bactéries, on peut la trouver en de nombreux sites et elle intervient souvent comme marqueur protégeant l'ADN de l'action d'enzymes de restriction sensibles à la méthylation ;
- chez les plantes, on la trouve dans les séquences CpG, CpHpG et CpHpH (où H = A, C ou T) ;
- chez les mycètes et les animaux, on la trouve essentiellement dans les dinucléotides CpG ; la plupart des eucaryotes ne méthylent qu'un faible pourcentage de ces sites, mais de 70 à 80 % des cytosines de dinucléotides CpG peuvent être méthylés chez les mammifères[3].
La désamination de la 5-méthylcytosine donne de la thymine, tandis que la désamination spontanée de la cytosine forme de l'uracile. Ce dernier, lorsqu'il est présent dans l'ADN, est reconnu et éliminé par les enzymes de réparation de l'ADN, tandis que la thymine est normalement présente dans l'ADN, de sorte que la conversion de la 5-méthylcytosine (m5C) en thymine (T) ne peut être détectée simplement. De surcroît, cette conversion peut être mutagène, car la m5C s'apparie avec la guanine (G) tandis que T s'apparie avec l'adénine (A). Par ailleurs, la désamination catalysée de la 5-méthylcytosine par les cytosines désaminases de la famille des APOBEC pourrait avoir des effets bénéfiques sur de nombreux processus cellulaires ainsi que sur le développement de ces cellules[4]. Les effets de la désamination de la 5-hydroxyméthylcytosine demeurent en revanche moins compris.
Notes et références
[modifier | modifier le code]- Masse molaire calculée d’après « Atomic weights of the elements 2007 », sur www.chem.qmul.ac.uk.
- (en) Vincent Colot et Jean-Luc Rossignol, « Eukaryotic DNA methylation as an evolutionary device », BioEssays, vol. 21, no 5, , p. 402-411 (PMID 10376011, DOI 10.1002/%28SICI%291521-1878%28199905%2921:5%3C402::AID-BIES7%3E3.0.CO;2-B, lire en ligne)
- (en) Kamel Jabbari et Giorgio Bernardi, « Cytosine methylation and CpG, TpG (CpA) and TpA frequencies », Gene, vol. 333, , p. 143-149 (PMID 15177689, DOI 10.1016/j.gene.2004.02.043, lire en ligne)
- (en) Richard Chahwan, Sandeep N. Wontakal et Sergio Roa, « Crosstalk between genetic and epigenetic information through cytosine deamination », Trends in Genetics, vol. 26, no 10, , p. 443-448 (PMID 20800313, DOI 10.1016/j.tig.2010.07.005, lire en ligne)


 French
French Deutsch
Deutsch