Base azotée — Wikipédia
Les bases azotées, ou bases nucléiques, ou bases nucléotidiques, voire nucléobases, sont des composés organiques azotés présents dans les acides nucléiques sous forme de nucléotides dans lesquels elles sont liées à un ose, le ribose dans le cas de l'ARN et le désoxyribose dans le cas de l'ADN. En génétique, on les désigne souvent simplement comme les bases des acides nucléiques. Elles jouent un rôle déterminant dans le stockage, l'expression et la transmission de l'information génétique en raison de leur aptitude particulière à s'apparier pour former des structures complémentaires. Sous forme de nucléotides, elles sont également indispensables au métabolisme énergétique des cellules, et l'une d'entre elles, l'adénine, intervient dans de nombreux processus biochimiques essentiels en formant des coenzymes. Elles sont désignées comme bases en référence à leurs propriétés acido-basiques caractérisées lors de leur découverte, mais leur nature basique n'intervient pas particulièrement dans leur fonction biologique fondamentale liée à l'hérédité.
Bases canoniques des acides nucléiques
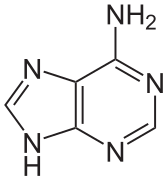
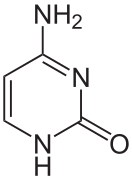
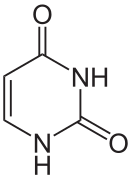
[modifier | modifier le code]Les cinq bases principales, ou canoniques, sont l'adénine, la cytosine, la guanine, la thymine et l'uracile, respectivement symbolisées par A, C, G, T et U. Parmi ces cinq bases, A, C, G et T sont les quatre bases de l'ADN, tandis que A, C, G et U sont les quatre bases de l'ARN. La thymine et l'uracile sont semblables à la différence près que la thymine possède un groupe méthyle sur l'atome de carbone 5. La guanine et l'adénine appartiennent à la famille des purines, qui sont symbolisées par R et sont des composés bicycliques comprenant deux hétérocycles à cinq et six atomes, tandis que la cytosine, la thymine et l'uracile sont des pyrimidines, qui sont symbolisées par Y et sont des composés monocycliques comprenant un hétérocycle à six atomes. Il existe par ailleurs d'autres bases, qui n'entrent pas dans la constitution des acides nucléiques et sont dites bases non canoniques.
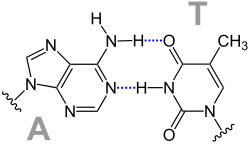
Dans la double hélice d'ADN, les bases s'apparient au moyens de liaisons hydrogène qui maintiennent les deux brins antiparallèles ensemble, une base purique s'appariant à une base pyrimidique. La cytosine et la guanine forment une paire de bases unies par trois liaisons hydrogène, tandis que l'adénine et la thymine forment une paire de bases unies par deux liaisons hydrogène.
L'analyse des séquences d'acides nucléiques rend parfois nécessaire de représenter une ou plusieurs bases en fonction de caractéristiques particulières. À cette fin, l'IUBMB a défini une nomenclature précise :
| Symbole[1] | Description | Bases représentées | ||||
|---|---|---|---|---|---|---|
| A | adénine | A | 1 | |||
| C | cytosine | C | ||||
| G | guanine | G | ||||
| T | thymine | T | ||||
| U | uracile | U | ||||
| W | weak | A | T | 2 | ||
| S | strong | C | G | |||
| M | amino | A | C | |||
| K | keto | G | T | |||
| R | purine | A | G | |||
| Y | pyrimidine | C | T | |||
| B | pas A (B vient après A) | C | G | T | 3 | |
| D | pas C (D vient après C) | A | G | T | ||
| H | pas G (H vient après G) | A | C | T | ||
| V | pas T (V vient après T et U) | A | C | G | ||
| N | any base | A | C | G | T | 4 |
Nucléosides et nucléotides
[modifier | modifier le code]Nomenclature
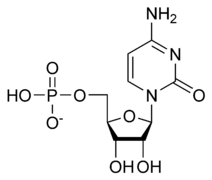
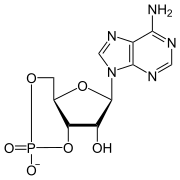
[modifier | modifier le code]La liaison d'une base nucléique à l'atome de carbone 1’ d'une molécule de ribose ou de désoxyribose donne un nucléoside, plus précisément un ribonucléoside (avec du ribose) ou un désoxyribonucléoside (avec du désoxyribose) ; la liaison d'un groupe phosphate à l'atome de carbone 5’ d'un nucléoside donne un nucléotide, plus précisément un ribonucléotide ou un désoxyribonucléotide.
- Nucléotides, nucléosides et bases nucléiques
Ci-dessous sont représentés en haut les ribonucléotides à guanine, cytosine, adénine et uracile constituant l'ARN, et en bas les désoxyribonucléotides à guanine, cytosine, adénosine et thymine constituant l'ADN.
Métabolisme
[modifier | modifier le code]Les bases nucléiques sont synthétisées dans les cellules sous forme de nucléotides, et plus particulièrement de ribonucléotides, c'est-à-dire liées à un résidu de ribose-5-phosphate.
Métabolisme des purines
[modifier | modifier le code]La formation de phosphoribosylpyrophosphate (PRPP, ou 5-phospho-α-D-ribose-1-diphosphate) par la PRPP synthétase est l'étape régulatrice clé de la biosynthèse des purines. Cette enzyme est activée par le phosphate inorganique et inhibée par les nucléotides puriques. Le PRPP est ensuite converti en inosine monophosphate (IMP), nucléotide dont la base est l'hypoxanthine et qui est formé à partir du PRPP au terme de plus d'une dizaine de transformations chimiques. Enfin, les autres nucléotides puriques sont formés à partir de l'IMP[2] :
- la guanosine monophosphate (GMP) est formée via la xanthosine monophosphate (XMP), sous l'action successive de l'IMP déshydrogénase et de la GMP synthase ;
- l'adénosine monophosphate (AMP) est formée via l'adénylosuccinate sous l'action successive de l'adénylosuccinate synthase et de l'adénylosuccinate lyase.
Diverses enzymes interviennent pour dégrader les nucléotides puriques. Ce sont tout d'abord des nucléotidases, qui hydrolysent les nucléotides en libérant le groupe phosphate et le nucléoside correspondant. Ce dernier est ensuite à son tour hydrolysé par une purine nucléoside phosphorylase afin de libérer le ribose-1-phosphate et la base correspondant au nucléoside : adénine dans le cas de l'adénosine, guanine dans le cas de la guanosine et hypoxanthine dans le cas de l'inosine. Cette dernière peut être formée à partir d'adénosine sous l'action de l'adénosine désaminase, ou à partir de l'AMP sous l'action de l'AMP désaminase pour donner de l'IMP, clivé par la suite en phosphate et en inosine par une nucléotidase.
Métabolisme des pyrimidines
[modifier | modifier le code]La biosynthèse des pyrimidines commence par la formation de carbamyl-phosphate sous l'action de la carbamyl-phosphate synthétase II[3]. Le carbamyl-phosphate est ensuite converti en acide carbamylaspartique sous l'action de l'aspartate carbamoyltransférase, puis en acide 4,5-dihydroorotique sous l'action de la dihydroorotase, puis en acide orotique sous l'action de la dihydroorotate déshydrogénase. Ce dernier est converti en nucléotide sous forme d'orotidine monophosphate (OMP) sous l'action de l'orotate phosphoribosyltransférase, qui utilise du phosphoribosylpyrophosphate comme substrat. Enfin, l'uridine monophosphate (UMP) est formée à partir de l'OMP par l'orotidine-5'-phosphate décarboxylase. Chez les mammifères, l'orotate phosphoribosyltransférase et l'orotidine-5'-phosphate décarboxylase forment une seule enzyme, l'uridine monophosphate synthétase.
L'UMP est ensuite phosphorylé successivement en uridine diphosphate (UDP) et en uridine triphosphate (UTP) successivement par l'uridine cytidine kinase 2 et une nucléoside diphosphate kinase, puis l'UTP est converti en cytidine triphosphate (CTP) par la CTP synthase.
Voies de sauvetage des nucléotides
[modifier | modifier le code]Dans la mesure où tous les tissus ne sont pas capables de synthétiser les nucléotides de novo, il existe des voies de sauvetage des nucléotides qui interviennent dans les cellules pour produire des nucléotides à partir de leurs produits de dégradation. Ainsi, des phosphoribosyltransférases forment des nucléotides monophosphates à partir de bases puriques et de phosphoribosylpyrophosphate (PRPP). Il existe deux types de phosphoribosyltransférases : l'adénine phosphoribosyltransférase (APRT), qui forme de l'AMP à partir d'adénine, et l'hypoxanthine-guanine phosphoribosyltransférase (HGPRT), qui forme de l'IMP et de la GMP à partir d'hypoxanthine et de guanine. Une déficience en HGPRT est responsable du syndrome de Lesch-Nyhan.
Rôle comme constituants de cofacteurs enzymatiques
[modifier | modifier le code]Les bases nucléiques — et particulièrement l'adénine — interviennent, sous forme de nucléosides et de nucléotides, dans de nombreux processus métaboliques indépendants de leur rôle de support de l'information génétique :
- sous la forme d'adénosine triphosphate (ATP) et de guanosine triphosphate, elles interviennent ainsi dans des processus aussi variés que la phosphorylation au niveau du substrat, la phosphorylation oxydative dans le cadre de la respiration cellulaire, la photophosphorylation dans le cadre de la photosynthèse, ou encore dans la transduction de signal (comme précurseur de l'AMP cyclique) ;
- Adénosine triphosphate (ATP)
- Guanosine triphosphate (GTP)
- AMP cyclique (AMPc)
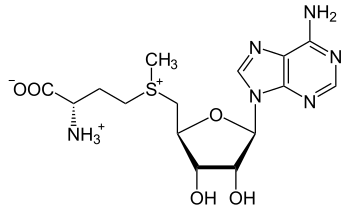
- sous la forme de dérivés de l'adénosine ou de l'adénosine monophosphate (AMP), elles interviennent dans un grand nombre de réactions catalysées par des enzymes en tant que coenzymes, par exemple comme constituants de la coenzyme A, du NAD+/NADH, du NADP+/NADPH, de la FAD/FADH2, ou encore de la S-adénosylméthionine (SAM).
- Coenzyme A (CoA)
- S-Adénosyl-L-méthionine (SAM)
Bases modifiées
[modifier | modifier le code]Outre les nucléosides à guanine, cytosine, adénine, thymine et uracile, il existe dans les ADN et ARN des nucléosides modifiés après leur incorporation dans les acides nucléiques. Dans l'ADN, la base modifiée la plus courante est la 5-méthylcytosine, dont le nucléoside est la 5-méthylcytidine (m5C). Dans l'ARN, et particulièrement les ARN de transfert, les nucléosides les plus fréquents sont la pseudouridine (Ψ), la dihydrouridine (DHU), l'inosine (I) et la 7-méthylguanosine (m7G).
- 5-Méthylcytidine (m5C)
- Pseudouridine (Ψ)
- Dihydrouridine (DHU)
- Inosine (I)
- 7-Méthylguanosine (m7G)
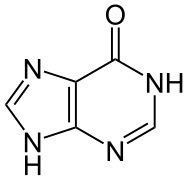
La xanthine et l'hypoxanthine, dont les nucléosides correspondants sont la xanthosine et l'inosine, sont formées en présence de mutagènes à partir de la guanine et de l'adénine respectivement[4]. De façon semblable, la désamination de la cytosine forme de l'uracile, ce qui est potentiellement mutagène dans la mesure où l'uracile peut s'apparier avec l'adénine alors que la cytosine s'apparie avec la guanine. Également, le même phénomène peut survenir avec la 5-méthylcytosine, générant la thymine par la désamination, qui s'apparie subséquemment à l'adénine plutôt qu'à la guanine.
Présence dans les chondrites carbonées
[modifier | modifier le code]Une étude financée par la NASA et publiée en 2011[5] a mis en évidence la présence d'un éventail de bases nucléiques dans les météorites de type chondrites carbonées, notamment l'adénine, la guanine, la xanthine, l'hypoxanthine et la purine, ainsi que deux bases absentes des systèmes biologiques connus : la 2,6-diaminopurine et la 6,8-diaminopurine[6].
Notes et références
[modifier | modifier le code]- ↑ (en) Nomenclature Committee of the International Union of Biochemistry (NC-IUB), « Nomenclature for Incompletely Specified Bases in Nucleic Acid Sequences », sur IUBMB, (consulté le ).
- ↑ (en) « AMP and GMP Biosynthesis », sur le site de l'IUBMB, (consulté le ).
- ↑ (en) « Pyrimidine Biosynthesis », (consulté le ).
- ↑ (en) T. Nguyen, D. Brunson, C. L. Crespi, B. W. Penman, J. S. Wishnok et S. R. Tannenbaum, « DNA damage and mutation in human cells exposed to nitric oxide in vitro », Proceedings of the National Academy of Sciences of the United States of America, vol. 89, no 7, , p. 3030-3034 (PMID 1557408, PMCID 48797, DOI 10.1073/pnas.89.7.3030, JSTOR 2359027, Bibcode 1992PNAS...89.3030N, lire en ligne)
- ↑ (en) Michael P. Callahan, Karen E. Smith, H. James Cleaves II, Josef Ruzicka, Jennifer C. Stern, Daniel P. Glavin, Christopher H. House et Jason P. Dworkin, « Carbonaceous meteorites contain a wide range of extraterrestrial nucleobases », Proceedings of the National Academy of Sciences of the United States of America, vol. 108, no 34, , p. 13995-13998 (PMID 21836052, PMCID 3161613, DOI 10.1073/pnas.1106493108, Bibcode 2011PNAS..10813995C, lire en ligne)
- ↑ (en) Aaron S. Burton, Jennifer C. Stern, Jamie E. Elsila, Daniel P. Glavin et Jason P. Dworkin, « Understanding prebiotic chemistry through the analysis of extraterrestrial amino acids and nucleobases in meteorites », Chemical Society Reviews, vol. 41, no 16, , p. 5459-5472 (PMID 22706603, DOI 10.1039/C2CS35109A, lire en ligne)
Liens externes
[modifier | modifier le code]- Ressource relative à la santé :
- Notice dans un dictionnaire ou une encyclopédie généraliste :


 French
French Deutsch
Deutsch