Immunohistochimie — Wikipédia
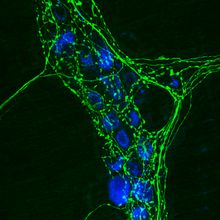
L'immunohistochimie (IHC) est une méthode de localisation de protéines dans les cellules d'une coupe de tissu, par la détection d'antigènes au moyen d'anticorps.
L'immunohistochimie exploite le fait qu'un anticorps se lie spécifiquement à des antigènes dans les tissus biologiques. Les anticorps peuvent être d'origine polyclonale ou monoclonale, les anticorps monoclonaux étant plus spécifiques par essence.
L'immunohistochimie tire son nom de la racine « immuno », en référence à l'anticorps utilisé dans la procédure, du mot « chimie » et du mot « histologie » qui évoque l'étude de tissus biologiques (à comparer à l'immunocytochimie).
Un couple anticorps-antigène peut être visualisé de plusieurs façons. Dans la plupart des cas, l'anticorps est conjugué à une enzyme (ex : peroxydase) qui peut catalyser une réaction de production de couleur (ex. : coloration immunoperoxydase). Les anticorps peuvent aussi être marqués par un fluorochrome (ex. : FITC, Rhodamine, Texas Red ou Alexa Fluor) : voir immunofluorescence.
Utilisation pour le diagnostic de cancers
[modifier | modifier le code]L'immunohistochimie est largement utilisée pour le diagnostic et/ou le suivi de cancers par détection de cellules anormales telles que celles trouvées dans les tumeurs cancéreuses.
Des marqueurs spécifiques sont ainsi aujourd'hui connus pour divers cancers :
- antigène carcino-embryonnaire (CEA) : utilisé en cas de cancer du côlon ;
- CD15 et CD30 : utilisés pour la maladie de Hodgkin ;
- alpha-fœtoprotéine : en cas de carcinome hépatocellulaire ;
- CD117 : en cas de tumeur stromale gastro-intestinale ;
- Ki-67 : un des indices de prolifération tumorale.
Ces marqueurs moléculaires sont caractéristiques de certains événements cellulaires tels que la prolifération ou la mort cellulaire (apoptose).
L'IHC est aussi largement utilisée en recherche fondamentale pour comprendre la distribution et la localisation de biomarqueurs et de protéines différentiellement exprimés dans les différentes parties d'un tissu biologique.
Détail de la méthode
[modifier | modifier le code]Le tissu est placé sur un support solide. L'anticorps est ajouté à la préparation de tissu, fixant spécifiquement la protéine à détecter sur ce tissu. L'ajout d'un anticorps primaire lié à un système de détection (enzyme liée à l'anticorps, dont la mise en présence du substrat provoque une réaction colorée (peroxydase) ou fluorescente (Rhodamine)), provoquant un signal visible à l'œil nu, ou par des techniques de microscopie et de spectrophotométrie.
Il existe principalement deux méthodes concernant l'immunohistochimie et la détection d'antigènes au sein des tissus biologiques :
- la méthode directe ;
- la méthode indirecte.
La « méthode directe » est une méthode de coloration en une seule étape. Elle utilise un anticorps marqué (c.-à-d. antisérum conjugué par FITC) qui réagit directement avec les antigènes présents dans les sections tissulaires. Un seul anticorps étant utilisé, la procédure est courte et rapide. Elle manque cependant de sensibilité en raison d'une faible amplification du signal, et est rarement utilisée depuis l'introduction de la méthode indirecte.
La « méthode indirecte » emploie un anticorps principal non-marqué (première couche) qui réagit avec les antigènes au sein du tissu, un anticorps secondaire marqué (seconde couche) qui réagit avec l'anticorps principal. L'anticorps secondaire doit être dirigé contre l'immunoglobuline G de l'anticorps principal. Cette méthode présente une meilleure sensibilité grâce à l'amplification du signal permise par plusieurs réactions de l'anticorps secondaire à différents sites antigéniques de l'anticorps principal. La seconde couche d'anticorps peut être marquée avec un colorant fluorescent tel que le FITC, la rhodamine ou le Texas Red, ce qui s'appelle la méthode par immunofluorescence indirecte. La seconde couche d'anticorps peut aussi être marquée par une enzyme telle que la peroxidase, la phosphatase alcaline ou la glucose oxydase. Cette dernière variante est appelée la méthode par immunoenzyme indirecte. Offrir un substrat à ces enzymes permet d'établir la présence de l'enzyme (et donc de l'anticorps) d'une manière quantifiable.
Protocole d'une méthode d'immunohistochimie à l'institut Bergonié
[modifier | modifier le code]Protocole : Mise au point de l’anticorps PEA3
[modifier | modifier le code]L’anticorps PEA3 est utilisé pour rechercher la protéine ETV4 qui est exprimé en grand nombre dans le sarcome à cellules rondes d’Ewing.
Le sarcome à cellules rondes d’Ewing est un cancer très agressif qui s’attaque principalement aux os et aux tissus mous chez des personnes assez jeunes. Il est lié à une translocation* au niveau des gènes CIC – DUX4, ce qui va produire une surexpression de la protéine ETV4.
PEA3 est un anticorps de souris monoclonal*. C’est-à-dire que l’on a injecté à la souris, l’antigène humain que l’on recherche (ETV4) dans le sarcome à cellules rondes d’Ewing, pour créer une réaction immunitaire chez la souris et pour qu’elle puisse produire les anticorps qui vont nous servir pour l’immunohistochimie.
- Lecture de la fiche technique
- Espèce chez laquelle est fabriqué l’anticorps (Source/Purification)
- Stockage (Frigo/Congélateur)
- Lot (aliquote* ou pas)
- Volume d’anticorps disponible
- Dilution (Soit proposé dans la fiche technique, soit se rapporter à un article publié)
- Réhydratation
Tout d’abord, pour réaliser une réaction antigène/anticorps, il faut réhydrater les tissus paraffinés sur la lame. Donc faire les étapes inverse où l’on met le tissu dans de la paraffine.
- 4 min dans chaque bain
- Xylène* (3 bains)
- Alcool absolu (2 bains)
- Alcool 90 °C (1 bain)
- Alcool 70 °C (1 bain)
- Plonger la lame immédiatement dans un récipient d’eau (pour que les tissus réhydratés ne sèchent pas), et faire rincer pendant 3 min en faisant couler l’eau du robinet dans le récipient.
- Plonger la lame dans un récipient d’eau distillée
- Prétraitement
- Diluer le pH9 au 1/10 (50 ml de tampon + 500 ml d’eau distillée) dans une éprouvette de 500 ml. Mettre la lame dans le pH9 dilué.
- Faire chauffer au micro-onde « programmé sur vapeur », la lame dans le pH9 dilué, pendant 30 min en ne fermant pas totalement le bocal (pour que la vapeur puisse s’échapper).
- Attendre que la solution pH9 dans le bocal refroidisse pendant 20 min.
- Rincer la lame dans un récipient d’eau pendant 3 min.
- Faire une délimitation des tissus, à l’aide d’un marqueur hydrophobe.
- Déposer de H2O2 (permet de neutraliser les peroxydases endogènes (dans le tissu) en vue de la coloration au DAB [peroxydases oxydent DAB produit coloré]) jusqu’à remplir le cadrant de délimitation et laisser reposer 10 min dans une chambre humide (car anticorps PEA3 agit de nuit).
- Pendant ce temps faire une dilution adaptée de l’anticorps PEA3.
- Rincer la lame dans un récipient d’eau.
- Mettre la lame dans un récipient d’eau distillée.
- Faire tremper la lame dans un récipient contenant un tampon de réaction (tampon de lavage qui permet à l’anticorps de mieux se répartir sur la lame car il a un effet «mouillant») pendant 5 min.
- Déposer l’anticorps (permet la fixation spécifique de l’anticorps (de souris) sur la protéine ETV4 [marquage nucléaire]) dans le cadrant de délimitation à l’aide d’une pipette et faire reposer la nuit au frigo.
- Révélation
- Rincer la lame avec du tampon de réaction.
- Laisser la lame 3 min dans un récipient contenant du tampon de réaction.
- Mettre l’anticorps de chèvre Rabbit/Mouse (cette chaîne de dextranes sur laquelle est couplée à l’anticorps de chèvre anti lapin/souris et est fixée un grand nombre de peroxydase, permet la fixation d'anticorps de chèvre sur les anticorps de souris [amplifie le signal donne une meilleure vue à l’observation microscopique]) pendant 30 min.
- Rincer avec du tampon de réaction.
- Laisser la lame dans un récipient contenant du tampon de réaction.
- Préparer la dilution de la DAB (1 ml de tampon DAB + 1 goutte de DAB) à l’aide d’une pipette compte-goutte.
- Mettre la révélation à la DAB (révélation de l’anticorps secondaire, par l’action enzymatique des peroxydases + DAB [produit coloré], donc localisé là où la protéine ETV4 est fixée sur l’anticorps primaire) sur le cadrant de délimitation et laisser reposer 8 min dans une boite hydratée cachée de la lumière.
- Rincer la lame dans un récipient d’eau.
- Mettre la lame dans un récipient d’eau distillée.
- Contre-coloration
- Plonger la lame pendant 5 à 10 secondes (si le tissu est grand) ou moins dans de l’hématoxyline (permet de colorer les noyaux/cytoplasme/membranes qui ne sont pas relevés par l’anticorps).
- Rincer à l’eau jusqu’à ce que le colorant parte.
- Mettre la lame dans un récipient contenant du tampon de réaction (virage bleu des noyaux/cytoplasme/membranes) pendant 2 min.
- Déshydrater (alcool 70 °C, alcool 90 °C, alcool absolu [x2], Xylène [x3]) et monter sous lamelle


 French
French Deutsch
Deutsch