Filogenia – Wikipédia, a enciclopédia livre
| Parte de uma série sobre |
| Evolução |
|---|
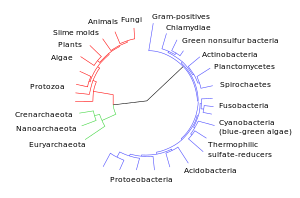 Diagrama da divergência dos grupos taxonómicos modernos em relação aos seus ancestrais comuns. |
| Tópicos fundamentais |
Em biologia, filogenia (ou filogênese) é o estudo da relação evolutiva entre grupos de organismos (por exemplo, espécies, populações), que é descoberto por meio de sequenciamento de dados moleculares e matrizes de dados morfológicos. O termo filogenética deriva dos termos gregos File (φυλή) e Filon (φῦλον), denotando "tribo" e "raça",[1] e o termo genético (γενετικός), denotando "em relação ao nascimento", da gênese (γένεσις) "origem" ou "nascimento".[2] O resultado dos estudos filogenéticos é a história evolutiva dos grupos taxonômicos, ou seja, sua filogenia.[3]
Taxonomia, a classificação, identificação e designação dos organismos, é ricamente baseada em informações da filogenia, mas são metodologicamente distintas.[4] Os campos de filogenia com sobreposição na taxonomia forma a sistemática filogenética - Uma metodologia cladística com características derivadas (sinapomorfias) usadas para encontrar o ancestral descendente na árvore (cladogramas e delimitam o taxon (clados).[5][6] Na sistemática biológica como um todo, as análises filogenéticas tornaram-se essenciais na pesquisa da árvore evolucionária da vida.
Construção de uma árvore filogenética
[editar | editar código-fonte]A evolução é considerada um processo de ramificação, onde as populações são alteradas ao longo do tempo e formam especiação em ramificações separadas, hibridizam juntas ou terminam em extinção. Isto pode ser visualizado em uma árvore filogenética.
O problema da filogenia é que os dados genéticos estão disponíveis apenas para taxons vivos e nos registros fósseis (dados osteométricos) contendo poucos dados e características morfológicas ambíguas.[7] Uma árvore filogenética representa uma hipótese da ordem dos eventos evolucionários ocorridos.
Cladística é o atual método de escolha para inferir árvores filogenéticas. Os métodos mais comumente usados para inferir filogenias incluem máxima parcimônia, semelhanças e MCMC baseada em inferência bayesiana. Fenética, popular no século XX, mas agora em grande parte obsoleto, usa Matriz de distâncias, baseada em métodos para a construção de árvores baseadas em semelhanças globais, que muitas vezes assumem relações filogenéticas aproximadas. Todos os métodos dependem de um modelo matemático explícito ou implícito que descreve a evolução das características observadas nas espécies e são normalmente utilizados pela Filogenética molecular, na qual os caracteres são alinhados em sequências de nucleótidos ou aminoácidos.
Agrupamento de organismos
[editar | editar código-fonte]
Existem alguns termos que descrevem a natureza de um agrupamento em tais árvores. Por exemplo, acredita-se que todas as aves e os répteis descendem de um único ancestral comum, por isso este agrupamento taxonômico (amarelo no diagrama) é chamado monofilético. Os "Répteis Modernos" (ciano no diagrama) constituem um agrupamento que contém um ancestral comum, mas não contém todos os descendentes desse ancestral (aves são excluídas); este é um exemplo de um grupo parafilético. Um agrupamento como animais de sangue quente que incluem mamíferos e aves (vermelho / laranja no diagrama) é chamado polifilético, porque não inclui o mais recente ancestral comum de seus membros.
Filogenia molecular
[editar | editar código-fonte]As conexões evolutivas entre organismos são representadas graficamente através de árvores filogenéticas. Devido ao fato de a evolução ocorrer durante longos períodos de tempo, algumas características não podem ser observadas diretamente, assim os biólogos devem reconstruir filogenias examinando marcadores genéticos comuns entre as espécies e inferir as relações evolutivas entre organismos. Fosseis podem ajudar com a reconstrução de filogenias, no entanto, os registros fósseis podem ter poucas informações para ajudar. Dessa forma, os biólogos normalmente estão restritos a análise de organismos atuais para identificar as suas relações evolutivas. As relações filogenéticas no passado foram reconstruídas olhando para fenótipos das características anatômicas. Hoje em dia, dados moleculares, que incluem sequências de proteínas e de DNA, são utilizados para a construção de árvores filogenéticas.[8]
O objetivo da Fundação Nacional da Ciência é a montagem da árvore de atividade da vida e resolver as relações evolutivas dos grandes grupos de organismos ao longo da história da vida, com pesquisas que muitas vezes envolvem grandes equipes de trabalho entre as instituições e disciplinas.
Teoria da recapitulação de Ernst Haeckel
[editar | editar código-fonte]
No final do século XIX, a Teoria da recapitulação de Ernst Haeckel ou a lei biogenética, foi amplamente aceita. Esta teoria foi muitas vezes expressa como "ontogenia recapitula a filogenia", isto é, o desenvolvimento de um organismo reflete exatamente o desenvolvimento evolutivo da espécie. A primeira versão de Haeckel desta hipótese é o embrião espelha os adultos ancestrais evoluído, e já foi rejeitada. A hipótese foi alterada para o desenvolvimento do embrião é o espelhamento de seus ancestrais evolutivos. A maioria dos biólogos modernos reconhecem numerosas conexões entre a ontogenia e filogenia, e as apresentam usando a biologia evolutiva do desenvolvimento ou mostrando como evidência de apoio a essa teoria. Donald I. Williamson sugeriu que as larvas e embriões representam adultos em outros táxons que foram transferidos por hibridação (a teoria da transferência das larvas).[9] A opinião de Williamson não representa o pensamento dominante na biologia molecular,[10] e há muitas evidências significativas contra a teoria da transferência das larvas.[11]
A transferência de genes
[editar | editar código-fonte]Em geral, os organismos podem herdar genes de duas formas: pela transferência vertical de genes e pela transferência horizontal de genes. A transferência gênica vertical é a passagem de genes de pais para filhos e a transferência horizontal de genes ou transferência lateral de genes ocorre quando os genes saltam entre organismos não relacionados (fenômeno comum em procariontes, sendo um exemplo a resistência adquirida aos antibióticos como resultado do intercâmbio de gene entre algumas bactérias e o desenvolvimento de múltiplas drogas resistentes para essas bactérias).
A transferência horizontal de genes tem complicado a determinação da filogenia de organismos e inconsistências na filogenia foram relatadas entre grupos específicos de organismos, dependendo dos genes usados para a construção da árvore evolutiva.
Carl Woese surgiu com a teoria dos três domínios da vida (archaea, eubactérias e Eucariotos) com base na sua descoberta de que os genes que codificam o RNA ribossômico são antigos e seus ancestrais transferiram genes, havendo assim transferência de genes com pouca ou nenhuma horizontalidade. Portanto, rRNAs são comumente recomendados como relógios moleculares para reconstruir filogenias.
Isso é particularmente útil para a filogenia de microorganismos, para a qual o conceito de espécie não se aplica e que são morfologicamente simples de classificar com base em características fenotípicas.
Amostragem de taxon e sinal filogenético
[editar | editar código-fonte]Devido ao desenvolvimento de técnicas avançadas de sequenciação em biologia molecular, tornou-se possível recolher grandes quantidades de dados (DNA ou sequências de aminoácidos) para inferir hipóteses filogenéticas. Não é raro encontrar estudos com matrizes de caracteres baseados em genomas mitocondriais (~ 16 mil nucleotídeos, em muitos animais). No entanto, tem sido proposto que isso aumenta do número de táxons na matriz do que aumenta o número de caracteres, porque quanto mais táxons mais robusta é a árvore filogenética resultante.[12]
Isto pode ser em parte devido à ruptura de atração de ramificações longas. Tem sido argumentado que esta é uma razão importante para incorporar dados de fosseis em filogenias sempre que possível. Naturalmente, os dados que incluem os táxons filogenéticos fóssil são geralmente baseadas na morfologia, em vez de dados de DNA. Usando simulações, Derrick Zwickl e David Hillis[13] descobriram que o aumento de amostragem de taxon em inferência filogenética tem um efeito positivo sobre a precisão das análises filogenéticas.
Outro fator importante que afeta a precisão da reconstrução da árvore consiste em saber se os dados analisados na verdade contêm um sinal filogenético útil, um termo que é usado geralmente para indicar se os organismos relacionados tendem a assemelhar-se com o seu material genético ou características fenotípicas.[14] Em última análise, não há nenhuma maneira de medir se uma hipótese filogenética é precisa ou não, a menos que as verdadeiras relações entre os táxons examinados já são conhecidas. O melhor resultado na sistemática pode ser alcançada na árvore com galhos bem suportados em evidências disponíveis.
Importância dos dados faltantes
[editar | editar código-fonte]Quanto mais dados estão disponíveis na construção de uma árvore, o mais precisa será a árvore resultante. A falta de dados é mais prejudicial do que ter menos dados, embora o seu impacto é maior quando a maioria dos dados em falta é de um pequeno número de táxons. Concentrar os dados faltantes em um pequeno número de caráter produz uma árvore mais robusta.[15]
O Papel dos fósseis
[editar | editar código-fonte]Muitas características morfológicas embrionárias e tecidos moles não podem ser fossilizados e torna a interpretação dos fósseis mais difíceis do que os táxons vivos. Isso às vezes torna difícil incorporar dados de fósseis à filogenia. No entanto, apesar destas limitações, a inclusão dos fósseis é de valor inestimável, para fornecer informações em áreas esparsas da árvore. Quebrando galhos longos e restringindo estados de caráteres intermediários. Assim os táxons fósseis contribuem tanto para resolver a árvore como os táxons modernos vivos.[16] Fósseis também pode restringir a idade das linhagens e demonstrar como uma árvore é consistente com o registro estratigráfico.[17]
Filogenias moleculares podem revelar a quantidade da diversificação, mas para definir a quantidade de táxons que se originaram ou se extinguiram, os dados devem ser trazidos dos fósseis.[18] As técnicas moleculares assumem uma quantidade constante de diversificação, que provavelmente e raramente seja verdade. Alguns casos, os pressupostos da interpretação do registro fóssil (por exemplo, um registro completo e imparcial) estão mais perto de ser verdadeiros que a hipótese de uma taxa constante, fazendo as descobertas dos fósseis mais precisas do que as reconstruções moleculares.[18]
Ponderação homoplasicas
[editar | editar código-fonte]Certos caracteres têm mais peso que outros e alguns caracteres devem ter menos peso na reconstrução de uma árvore.[19] Infelizmente, o único modo objetivo para determinar a importância é pela construção de uma árvore. Mesmo assim, os caracteres de ponderação homoplasicas conduzem a um melhor suporte nas árvores.[19] Refinamento adicional pode ser trazido por mudanças de ponderação e alteração em outras. Por exemplo, a presença de asas torácicas quase garante a colocação de um inseto entre os pterygota, embora as asas sejam frequentemente perdidas, a sua ausência não exclui o taxon do grupo.[20]
Ver também
[editar | editar código-fonte]Referências
- ↑ Liddell, Henry George; Robert Scott (1901). A Greek-English lexicon. Oxford: Clarendon Press. 1698 páginas
- ↑ Liddell, Henry George; Robert Scott (1901). A Greek-English lexicon. Oxford: Clarendon Press. 305 páginas
- ↑ «phylogeny». Biology online
- ↑ Edwards AWF; Cavalli-Sforza LL (1964). «Reconstruction of evolutionary trees». In: Heywood, Vernon Hilton; McNeill, J. Phenetic and Phylogenetic Classification. [S.l.: s.n.] pp. 67–76. OCLC 733025912.
Phylogenetics is that branch of life science, which deals with the study of evolutionary relation among various groups of organisms, through molecular sequencing data.
- ↑ Speer, Vrian (1998). «UCMP Glossary: Phylogenetics». UC Berkeley. Consultado em 22 de março de 2008
- ↑ E.O. Wiley, D. Siegel-Causey, D.R. Brooks, V.A. Funk. 1991. The Compleat Cladist: A Primer of Phylogenetic Procedures. Univ. Kansas Mus. Nat. Hist. (Lawrence, KS), Spec. Publ. No 19 online at Internet Archive
- ↑ Cavalli-Sforza, L. L.; Edwards, A. W. F. (1967). «Phylogenetic Analysis: Models and Estimation Procedures». Evolution. 21 (3): 550–70. JSTOR 2406616. doi:10.2307/2406616
- ↑ Pierce, Benjamin A. (17 de dezembro de 2007). Genetics: A conceptual Approach 3rd ed. [S.l.]: W. H. Freeman. ISBN 978-0-7167-7928-5
- ↑ Hybridization in the evolution of animal form and life-cycle | Zoological Journal of the Linnean Society | Oxford Academic
- ↑ John Timmer, "Examining science on the fringes: vital, but generally wrong", ARS Technica, 9 November 2009
- ↑ Hart, Michael W.; Grosberg, Richard K. (2009). «Caterpillars Did Not Evolve from Onychophorans by Hybridogenesis». Proceedings of the National Academy of Sciences. 106 (47): 19906–9. JSTOR 25593297. PMC 2785265
 . PMID 19880752. doi:10.1073/pnas.0910229106
. PMID 19880752. doi:10.1073/pnas.0910229106 - ↑ Wiens, John J. (2006). «Missing data and the design of phylogenetic analyses». Journal of Biomedical Informatics. 39 (1): 34–42. PMID 15922672. doi:10.1016/j.jbi.2005.04.001
- ↑ Zwickl, Derrick J.; Hillis, David M. (2002). «Increased Taxon Sampling Greatly Reduces Phylogenetic Error». Systematic Biology. 51 (4): 588–98. PMID 12228001. doi:10.1080/10635150290102339
- ↑ Blomberg, Simon P.; Garland, Theodore; Ives, Anthony R. (2003). «Testing for phylogenetic signal in comparative data: Behavioral traits are more labile». Evolution. 57 (4): 717–45. PMID 12778543. doi:10.1111/j.0014-3820.2003.tb00285.x
- ↑ Prevosti, Francisco J.; Chemisquy, María A. (2009). «The impact of missing data on real morphological phylogenies: Influence of the number and distribution of missing entries». Cladistics. 26 (3). 326 páginas. doi:10.1111/j.1096-0031.2009.00289.x
- ↑ Cobbett, Andrea; Wilkinson, Mark; Wills, Matthew (2007). «Fossils Impact as Hard as Living Taxa in Parsimony Analyses of Morphology». Systematic Biology. 56 (5): 753–66. PMID 17886145. doi:10.1080/10635150701627296
- ↑ Huelsenbeck, John P. (1994). «Comparing the Stratigraphic Record to Estimates of Phylogeny». Paleobiology. 20 (4): 470–83. JSTOR 2401230
- ↑ a b Quental, Tiago B.; Marshall, Charles R. (2010). «Diversity dynamics: Molecular phylogenies need the fossil record». Trends in Ecology & Evolution. 25 (8). 434 páginas. doi:10.1016/j.tree.2010.05.002
- ↑ a b Goloboff, Pablo A.; Carpenter, James M.; Arias, J. Salvador; Esquivel, Daniel Rafael Miranda (2008). «Weighting against homoplasy improves phylogenetic analysis of morphological data sets». Cladistics. 24 (5). 758 páginas. doi:10.1111/j.1096-0031.2008.00209.x
- ↑ Goloboff, Pablo A. (1997). «Self-Weighted Optimization: Tree Searches and Character State Reconstructions under Implied Transformation Costs». Cladistics. 13 (3). 225 páginas. doi:10.1111/j.1096-0031.1997.tb00317.x


 French
French Deutsch
Deutsch